구글은 여러 포털사이트 중 전 세계적으로 높은 인지도와 높은 시장 점유율을 가지고 있다. 이런 이유는 구글이 제공하는 여러 서비스(강력한 검색, 구글맵, 무료 이메일)들이 있기 때문이다. 그 중 구글에서 무료로 제공하는 구글맵 API를 이용한 웹에서 활용한 시각화법을 알아보고자 한다. 이 방법은 웹에서 구글맵을 이용하면 통계데이터나 위치기반 정보를 표현하는데 유리하다. 다시 말해 맵 위에 정보를 얹혀 사용자의 이해력을 향상시키고 직관적인 정보를 보여줄 수 있기 때문이다.
현재 구글에서 제공 중인 구글맵 자바스크립트 API 버전은 v3이다. v2도 사용은 되고 있지만 구글에서는 v3으로 마이그레이션하기를 권고하고 있다. 구글맵 API의 자세한 내용은 https://developers.google.com/maps/documentation/javascript/?hl=ko 에서 확인할 수 있다.
그럼 웹에 구글맵을 얹고 우리가 원하는 위치에 마커를 찍고 분포도에 따른 히트맵 표현 후 마지막으로 마커 개수에 따라 클러스트링 하는 방법을 알아보자.
시작은 간단하게...(웹에 맵 띄우기)
가장 먼저 심플하게 지도를 얹는 방법부터 알아보자. 아래는 전체 코드이며 앞으로 이 코드에서 소스를 추가하여 계속 응용 할 것이다.
줄 번호 보이기/숨기기
1 <!DOCTYPE html>
2 <html>
3 <head>
4 <title>google map</title>
5 <meta charset="utf-8">
6 <style>
7 html,body,
8 height: 100%;
9 margin: 0px;
10 padding: 0px
11 }
12 </style>
13 <script type="text/javascript" src="https://maps.googleapis.
com/maps/api/js?libraries=visualization&sensor=false"></script>
14 <script>
15 var map;
16 function initialize() {
17 var mapOptions = {
18 zoom : 7,
19 center : new google.maps.LatLng(37.5651,
126.98955), //서울
20 mapTypeId : google.maps.MapTypeId.ROADMAP
21 };
22 map = new google.maps.Map(document.getElementById
('map-canvas'), mapOptions);
23 }
24 google.maps.event.addDomListener(window, 'load', initialize);
25 </script>
26 </head>
27 <body>
28 <div id="map-canvas"></div>
29 </body>
30 </html>
맵을 띄우기 위한 가장 기본적인 라인은 13번이다. 여기서 구글에서 제공하는 API를 로드한다.
뒤에 붙는 파라미터 중 sensor는 필수사항이며, true or false를 선택할 수 있으며 의미하는 바는 사용자 위치를
확인하기 위해 센서를 사용할 것인지에 대한 명시이다. 우리는 위도, 경도 값을 이용하여 위치를 찾을 것이므로 false로 한다. 그
이외에 language=ko or en 등 언어를 설정 할 수도 있다. 설정하지 않을 시 어플리케이션의 기본언어 설정을
따른다. libraries=visualization은 마커를 사용하기 위한 라이브러리 옵션이다. 다음으로 24번 라인에 의해서
initialize함수를 호출하게 되며 이 함수에서는 초기 맵에 대해 설정을 한 후 맵을 로드하게 된다. 22번 라인에서
document에서 element id가 "map-canvas"라는 부분에 맵을 띄워라 라는 의미이다.
아래 이미지가 위 코드를 실행 하였을 때의 화면이다.
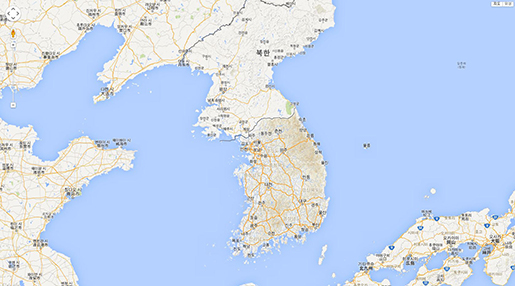 GeoCoder이용 마커표시(지역->위도경도변환)
GeoCoder이용 마커표시(지역->위도경도변환)
구글에서는 지역을 입력받아 그 지역의 위도와 경도를 반환해주는 geocoder를 제공하고 있다. 아래는 해당소스이며, 반환된 위도, 경도에 마커를 찍어 보자.
줄 번호 보이기/숨기기
1 <style>
2
3 position: absolute;
4 top: 5px;
5 left: 50%;
6 margin-left: -180px;
7 z-index: 5;
8 background-color:
9 padding: 5px;
10 border: 1px solid
11 }
12 </style>
13 .
14 .
15 .
16 var geocoder;
17 function codeAddress() {
18 geocoder = new google.maps.Geocoder();
19 var address = document.getElementById('address').value;
20 geocoder.geocode( { 'address': address}, function(results,
status) {
21 if (status == google.maps.GeocoderStatus.OK) {
22 var lat = results[0]['geometry']['location']
['lat']();
23 var lng = results[0]['geometry']['location']
['lng']();
24 alert(address+"의 위도는 " + lat + " 이며, 경도는
" + lng + " 입니다.");
25 map.setCenter(results[0].geometry.location);
26 var marker = new google.maps.Marker({
27 map: map,
28 position: results[0].geometry.location,
29 draggable:false,
30 animation:google.maps.Animation.DROP,
31 title:address
32 });
33 } else {
34 alert('Geocode was not successful for the following
reason: ' + status);
35 }
36 });
37 }
38 .
39 .
40 .
41 <body>
42 <div id="panel">
43 <input id="address" type="textbox" value="Seoul">
44 <input type="button" value="GO" onclick="
codeAddress()">
45 </div>
46 <div id="map-canvas"></div>
47 </body>
17번 라인의 codeAddress()함수에서 지역을 입력받아 위도, 경도로 변환하여 그 위치에 마커를 찍어주는 역할을 한다.
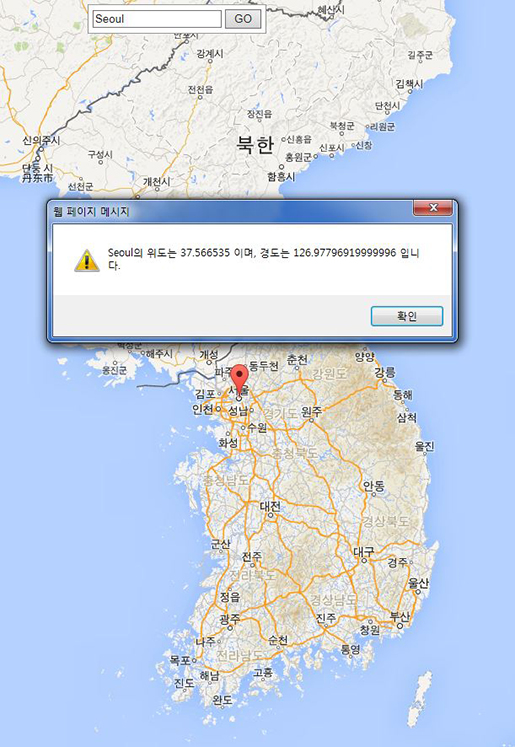
아래 소스처럼 조금 응용하면 다중마커를 찍히게 할 수 있다. 활용사례로는 지역별 감기발생현황등을 예로 들수 있다.
줄 번호 보이기/숨기기
1 function geocode() {
2 var addressList = new Array( '서울', '대구','광주','부산',
'포항','강릉','전주','광양','거제','김해','대전','울산','마산','수원','인천',
'군산','파주');
3 for(var j in addressList){
4 codeAddress(addressList[j]);
5 }
6 }
7 function codeAddress(address) {
8 geocoder = new google.maps.Geocoder();
9 geocoder.geocode( { 'address': address}, function (results,
status) {
10 .
11 .
12 .
13 }
실질적으로 활용을 하려면 관련 데이터가 데이터베이스화 되어 있거나 파일로 정형화가 되어 있어야 한다. 위 방법은 다중 마커를 보여주기 위해 임의로 배열을 이용하여 지역을 넣었다. 위 코드를 실행하면 아래와 같다.
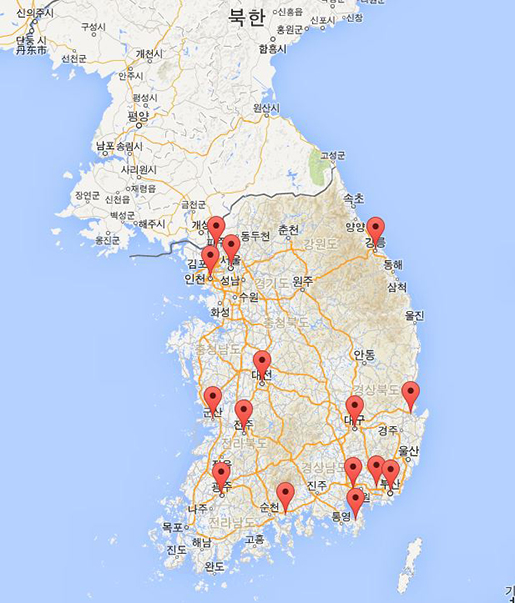
다만 다중마커를 위해 geocode를 빠른시간내 지속적으로 사용하게되면
geocode was not successful for the following reason over_query_limit 라는 에러가 발생하게 된다. 이를 해결하기 위해 다음코드를 추가할 수도 있다.
줄 번호 보이기/숨기기
1 if (status === google.maps.GeocoderStatus.OVER_QUERY_LIMIT) {
2 setTimeout(function() {
3 codeAddress(address);
4 }, 200);
5 }
만약 쿼리조회 중 "OVER_QUERY_LIMIT"라는 상태가 나타나면 0.2초간 대기 후 다시
쿼리를 조회하게 된다. 이렇게 되면 또 하나의 문제가 발생하게 된다. 마커를 찍는 시간이 마커의 개수에 비례하여 증가하게 된다는
것이다. 제일 좋은 방법은 지리적 정보를 데이터베이스화 하는 것이고, 추가되는 지리적 정보에 대해서만 geocode를 사용하여
결과를 DB에 추가 입력하는 방법이다.
HeatMap 표현
예를 들어 A라는 지역에 감기환자가 10명이 발생하였고, B라는 지역에 감기환자가 1명이 발생하였다고 하자. 그럴 경우 마커만으로는 정보를 보여주기에는 한계가 있다. 이를 해결하기 위해 가중치를 이용한 HeatMap을 표현할 수 있다. 방법은 아래와 같다.
줄 번호 보이기/숨기기
1 var heatmap;
2 var heatMapData = [];
3
4 function geocode(addressList) {
5 var addressList = new Array('서울','서울','서울','서울','서울','서울',
'서울','서울','대구','광주','부산','포항','강릉','전주','광양','거제','김해',
'대전','울산','마산','수원','인천','서울','대구','광주','부산','포항','강릉',
'전주','서울','서울','서울','광양','거제','김해','대전','울산','마산',
'서울','서울','수원','인천','군산','파주','서울','서울');
6 for ( var j in addressList) {
7 codeAddress(addressList[j]);
8 }
9 heatmap = new google.maps.visualization.HeatmapLayer({
10 data : heatMapData,
11 radius : 40,
12 dissipating : true
13 });
14 heatmap.setMap(map);
15 }
16 .
17 .
18 .
19 function codeAddress(address) {
20 geocoder = new google.maps.Geocoder();
21 geocoder.geocode({'address' : address}, function
(results, status) {
22 if (status == google.maps.GeocoderStatus.OK) {
23 var lat = results[0]['geometry']
['location']['lat']();
24 var lng = results[0]['geometry']
['location']['lng']();
25 map.setCenter(results[0].geometry.
location);
26 var marker = new google.maps.Marker({
27 map : map,
28 position : results[0].geomet
ry.location,
29 draggable : false,
30 animation : google.maps.
Animation.DROP,
31 title : address
32 });
33 var weightedLoc = {
34 location : new google.maps.
LatLng(lat, lng),
35 weight : 2 * 가중치(숫자)
36 };
37 heatMapData.push(weightedLoc);
38 } else {
39 alert('Geocode was not successful for
the following reason: '+ status);
40 }
41 });
42 }
먼저 HeatMap을
사용하기 위해 heatmap과 heatMapData를 선언을 한다. codeAddress함수내 33-36번 라인에서 가중치를
계산해주고, 계산된 가중치 값을 heatMapData에 담게된다. 5번 라인의 모든 지역에 대한 가중치가 heatMapData에
담기게 되고, 9-13번 라인에서 HeatMap 옵션을 정하고, 14번 라인에서 맵에 HeatMap을 뿌려 주게 된다.
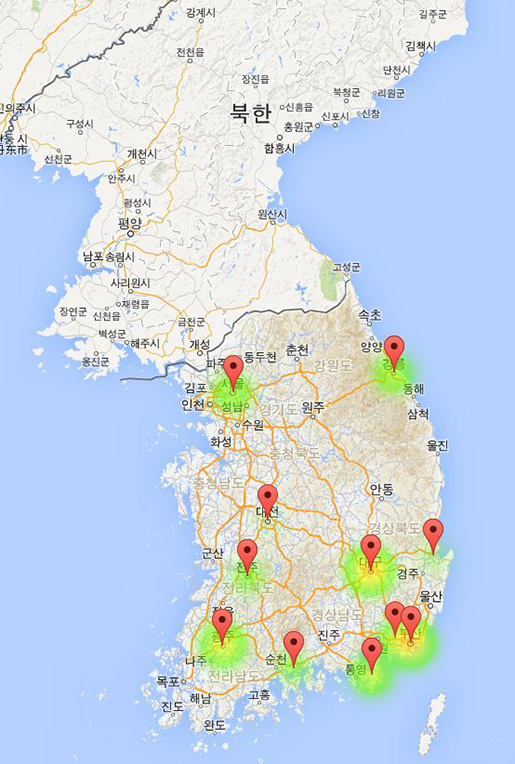 마커 클러스터링
마커 클러스터링
마커의 개수가 많아지면 시각적으로 복잡해 보이며, 화면에 마커를 뿌려 주는 시간이 마커개수에 비례하여 증가하게 된다. 이러한 문제를 해결하기 위해 구글맵은 클러스터링을 제공한다.
줄 번호 보이기/숨기기
1 <script type="text/javascript" src="http://google-maps-utility-
library-v3.googlecode.com/svn/trunk/markerclusterer/src/
markerclusterer.js"></script>
2
3 var markerCluster;
4 var markers = new Array();
5 function geocode() {
6 var addressList = new Array('서울','서울','서울','서울','서울','서울','서울','서울','대구','광주','부산','포항','강릉','전주','광양','거제','김해','대전','울산','마산','수원','인천','서울','대구','광주','부산','포항','강릉','전주','서울','서울','서울','광양','거제','김해','대전','울산','마산','서울','서울','수원','인천','군산','파주','서울','서울');
7 for(var j in addressList){
8 codeAddress(addressList[j]);
9 }
10 heatmap = new google.maps.visualization.HeatmapLayer({
11 data: heatMapData,
12 radius: 30,
13 dissipating: true
14 });
15 heatmap.setMap(map);
16
17 var clusterOptions = {
18 gridSize: 30,
19 minimumClusterSize: 2
20 };
21 markerCluster = new MarkerClusterer(map, markers,
clusterOptions);
22 }
23 .
24 .
25 .
26 function codeAddress(address) {
27 geocoder = new google.maps.Geocoder();
28 geocoder.geocode( { 'address': address}, function
(results, status) {
29 if (status == google.maps.GeocoderStatus.OK)
{
30 var lat = results[0]['geometry']
['location']['lat']();
31 var lng = results[0]['geometry']
['location']['lng']();
32 map.setCenter(results[0].geometry.
location);
33 var marker = new google.maps.Marker
({
34 map: map,
35 position: results[0].geometry
.location,
36 draggable:true,
37 animation:google.maps.Animat
ion.DROP,
38 title:address
39 });
40 markers.push(marker);
41
42 var weightedLoc = {
43 location: new google.maps.LatLng
(lat, lng),
44 weight: 2 * 가중치(숫자)
45 };
46 heatMapData.push(weightedLoc);
47 } else if (status === google.maps.Geocoder
Status.OVER_QUERY_LIMIT) {
48 setTimeout(function() {
49 codeAddress(address);
50 }, 200);
51 } else {
52 alert('Geocode was not successful for
the following reason: ' + status);
53 }
54 });
55 }
마커클러스터를 사용하기 위해 1번 라인에 클러스터 관련 자바스크립트파일(js format)을 로드한다. 그리고
markerCluster와 마커개수를 담을 수 있는 markers를 선언을 한다. 40번 라인에서 마커에 정보를 markers에
담는다. 17-20번 라인에서 마커클러스터 옵션을 설정 후 21번 라인에서 맵에 뿌린다. 그러면 gridSize에 값에 따라 맵을 그리드로 나눈 후 해당 그리드 안에 있는 마커들을 클러스터링 한다.
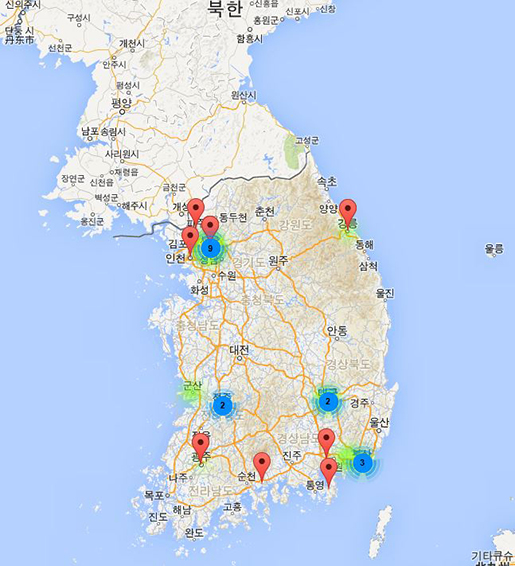
활용방안
지역적(국내외) 통계를 다루는 데이터가 있는 곳이면
활용 가능하다. 예를 들면 당사와 같이 생물정보를 전문적으로 다루는 곳이라면 바이러스와 같은 발생 지역에 대한 분포도를 보기위해
사용할 수 있고, 교통정보를 다루는 곳이라면 시시각각 차가 막히는 구간을 Heatmap으로 표현하는 방법으로 사용될 수
있을것이다.
지금까지 웹에서 구글맵을 활용하는 방법을 간단히 알아보았다. 위와 같은 방법을 잘 활용하여 사용자가 원하는 방향의 스마트한 웹을 구현하는 것이 개발자로서 가져야 할 책임과 임무라고 생각하며 이 글을 마친다.
작성자 : 데이터사이언스센터 통합개발실
최석문 주임 개발자
Posted by 人Co


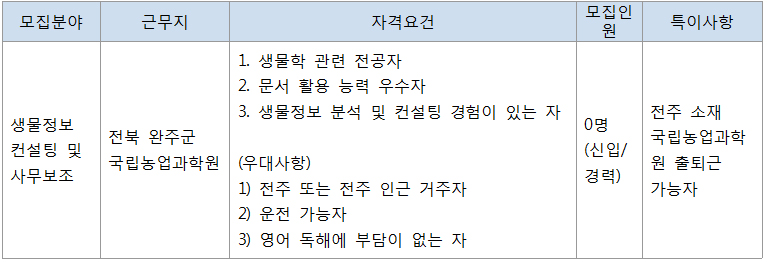
 人CoINTERNSHIP_지원서.doc
人CoINTERNSHIP_지원서.doc
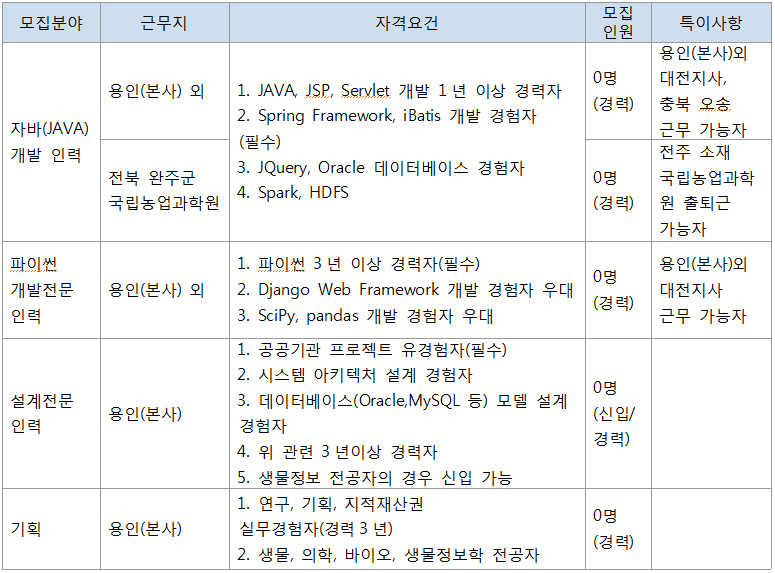
 입사지원서_2015.docx
입사지원서_2015.docx