Power with Simplicity
- Posted at 2016/09/07 13:21
- Filed under 제품소식
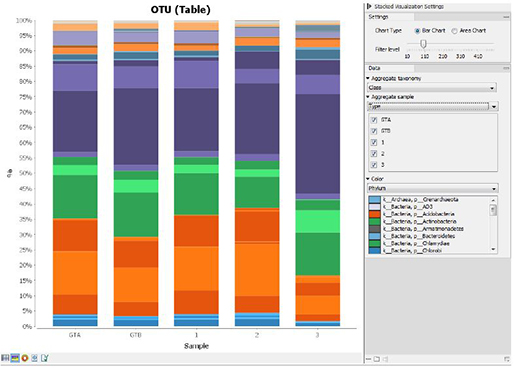
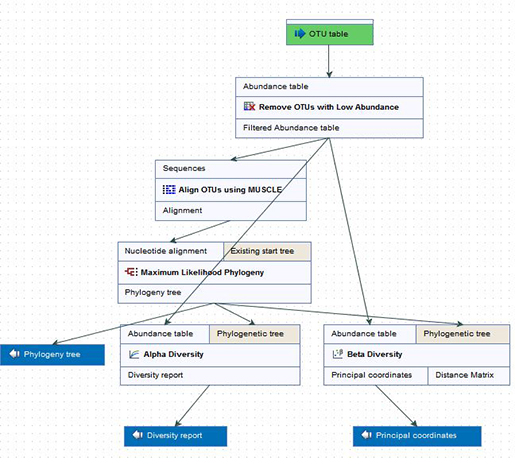
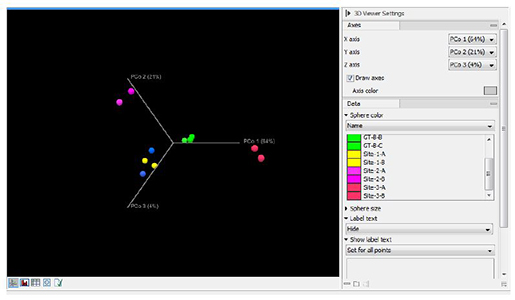
그럼 NGS 데이터를 이용하여 실제적으로 어떤 분석이 가능한지 살펴볼까요?
Next-Gen Sequencing
[Reference assembly]
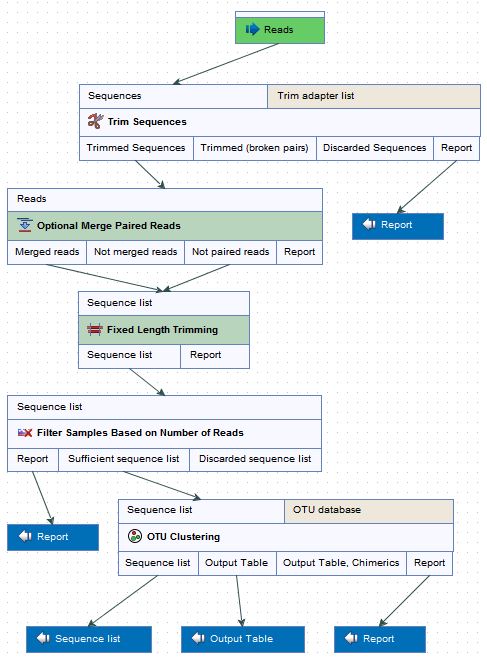
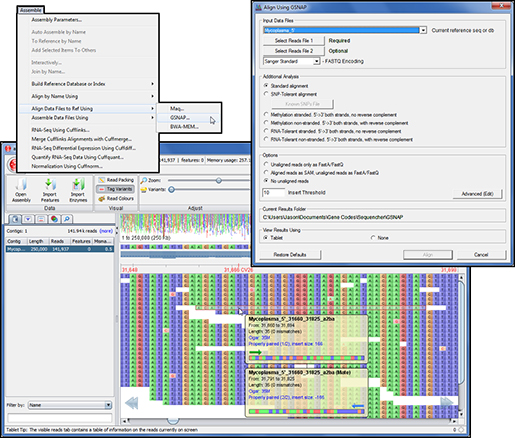
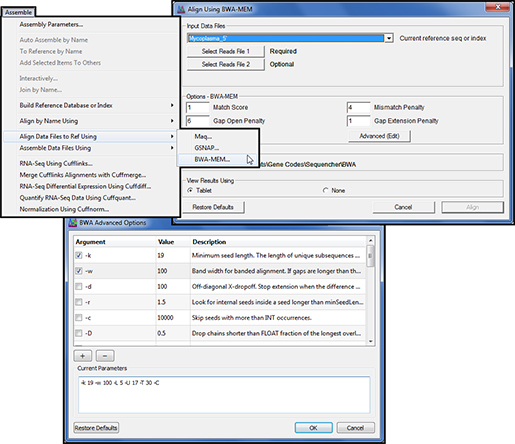
Sequencher에서는 NGS 데이터를 이용하여 reference assembly 시 이용하는 3개의 큰 알고리즘(Maq, GSNAP,
BWA-MEM)이 있습니다. Maq이나 GSNAP을 통해 assemgbly 분석을 진행하면 SNP 분석도 함께 가능하며, 그
결과 값은 Tablet이나 Maqview를 이용하여 확인할 수 있습니다. 커맨드라인으로 제공하던 BWA-MEM도 GUI를 통해
다양한 옵션값을 손쉽게 설정할 수 있습니다. GSNAP이나 BWA-MEM로부터 얻은 VCF 포맷의 variant 정보는
SAMtools를 이용하여 분석할 수 있습니다.
[De novo assembly]
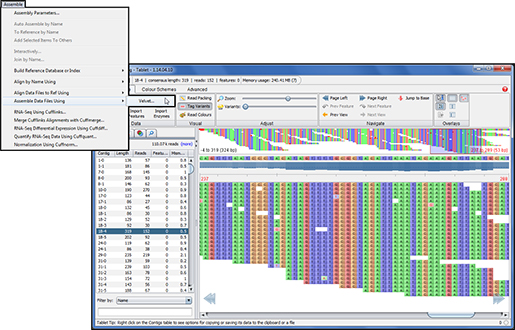
Reference 정보가 없는 de novo assembly의 경우에는 Velvet 알고리즘을 지원하고 있습니다. Velvet 또한 GUI를 제공함으로써, Tablet으로 결과값을 확인할 수 있고, 다양한 옵션값을 쉽게 설정할 수 있습니다.
[RNA-seq]
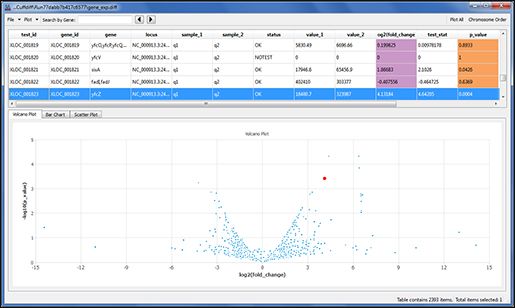
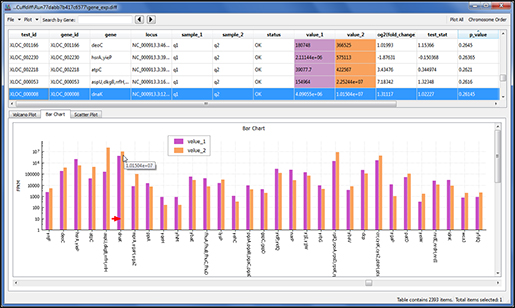
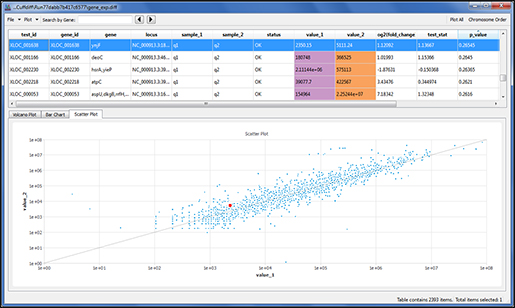
최신버전에서는 Differential Gene Expression(차등유전자발현) 연구를 위해 가장 많이 이용되는 RNA-seq 툴 중 하나인 Cufflinks를 플러그인으로 사용할 수 있습니다. Cufflinks 는 SAM 파일로부터 align된 reads를 가지고 GTF annotation 파일을 이용해 다시 align 하며, 다른 isoform과 transcript를 찾아줍니다. 이후 Cuffmerge를 통해 Cufflinks에서 나온 두 개의 transcript 파일을 하나의 transcript consensus 파일로 만들어 줍니다. 이 파일은 차등유전자발현 분석을 하는 Cuffdiff에 사용됩니다. Sequencher는 Cuffdiff에서 나오는 최종파일들(volcano plot, scatter plot, bar chart)을 다루며 발현 레벨에서 차이점을 그래픽으로 보여줍니다.
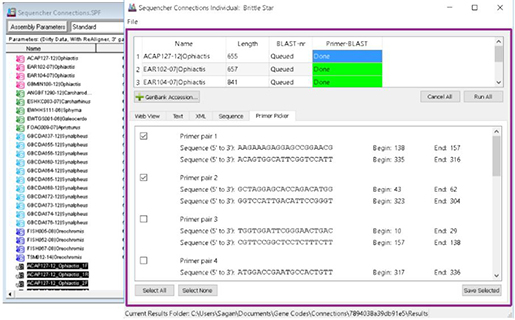
Connections
[BLAST & primer-BLAST]
Sequencher Connections는 Sequencher의 통합 웹 확장 툴이며, 이를 이용하여 2개 이상의 분석들을 동시에 진행할 수 있습니다. 다중 BLAST를 진행할 수 있어, 각 서열의 분석 결과를 실시간으로 빠르게 얻을 수 있습니다. 같은 서열로 다른 파라미터 조건을 주어 BLAST가 가능하며, 동시에 Local BLAST 까지도 수행할 수 있습니다. 또한 primer design을 위한 primer-BLAST를 할 수 있고, 해당 서열의 특정 영역을 확인 후 Sequencher Project에 예측된 primer를 저장할 수 있습니다. BLAST 검색 결과를 Web view 탭을 통해 뷰어할 수 있고, 이는 36시간 내에 다시 불러올 수 있으며, 그 이후로는 접근이 어렵습니다.
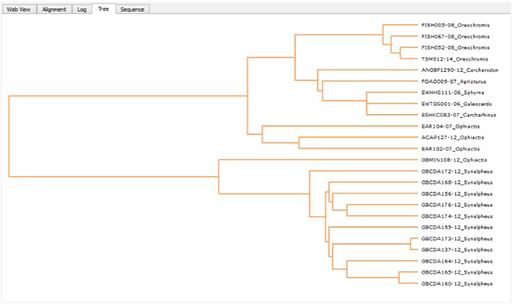
[MUSCLE alignment]
만일 여러 개의 서열로 그룹 분석을 하고 있다면, 다중서열정렬 알고리즘 중 가장 빠른 MUSCLE을 가지고 alignment를 할 수 있습니다. Sequencher Connections에서는 MUSCLE alignment를 다양한 옵션값으로 할 수 있고, alignment 이 후 phylogenetic tree도 생성할 수 있어 서열간의 유연관계도 확인할 수 있습니다.
이렇게 Sequencher에서는 NGS 분석까지 가능하도록 툴들이 확장되고 있습니다. 특히나 커맨드라인의 툴들을 사용하기 어려운 일반 생물학자들도 쉬운 인터페이스를 가진 Sequencher를 이용하여 NGS assembly를 진행할 수 있습니다. 그럼 Sequencher를 통해 NGS의 다양한 분석을 진행해 보세요.
송하나 주임 컨설턴트
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/220