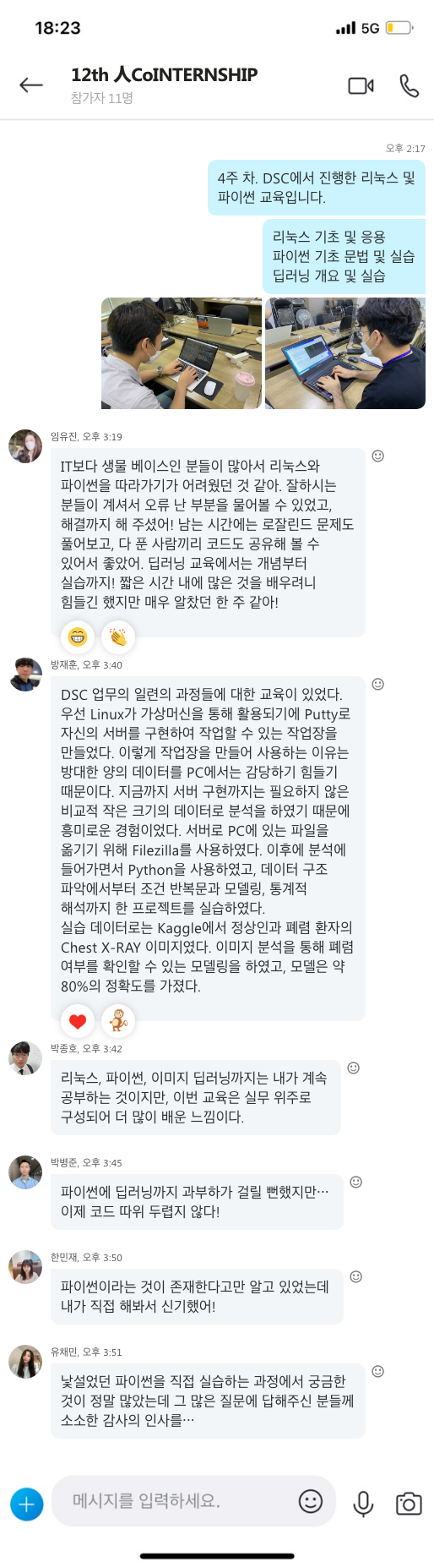
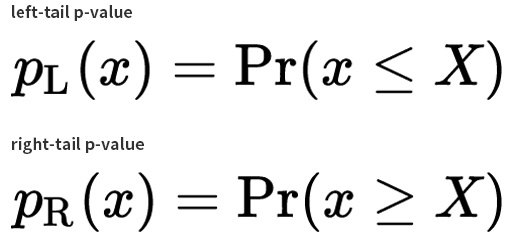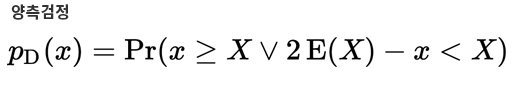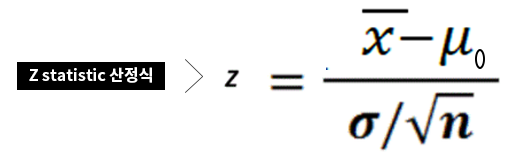지난해 12월부터 지금까지 전 세계 보건의료를 극심하게 뒤흔들고 있는 SARS-CoV-2의 유전체는 29.8kb 염기서열로 구성되어 있으며 27개의 단백질을 코딩하는 14개의 ORF(Open Reading Frame)를 가지고 있습니다.
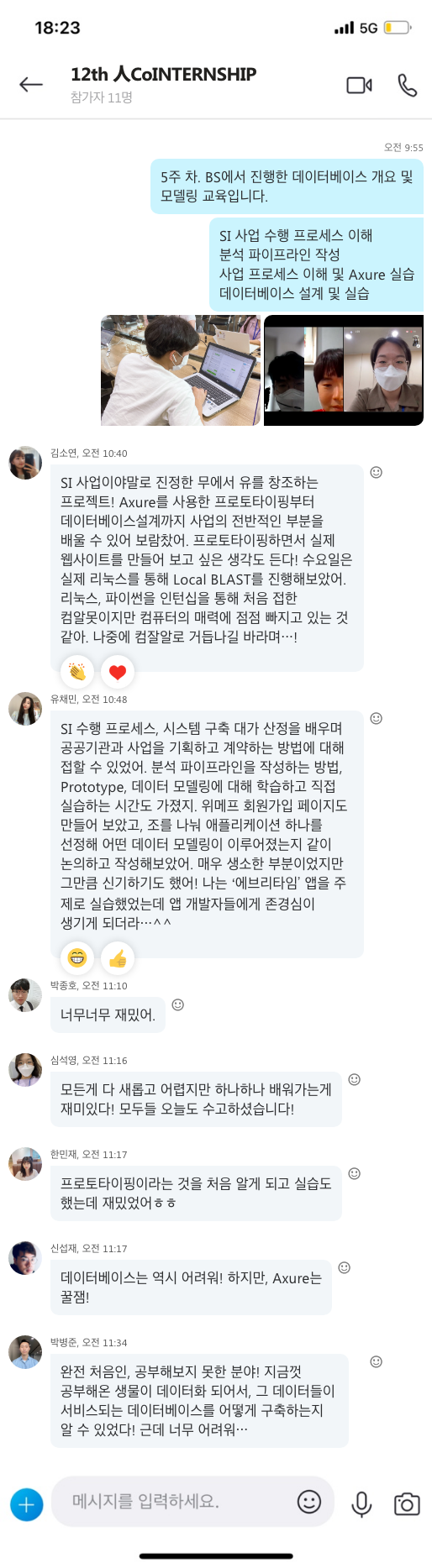
이번 SARS-CoV-2 관련한 환자 유래 바이러스 유전체 시퀀싱 데이터는 GISAID(https://www.gisaid.org/) 사이트에서 모두 공개하고 있습니다. 데이터베이스 내 유전체 계통분석 결과를 보면 바이러스 변이가 얼마나 빠르게 생겨나고 어떻게 확산되고 있는지 패턴을 확인할 수 있습니다.
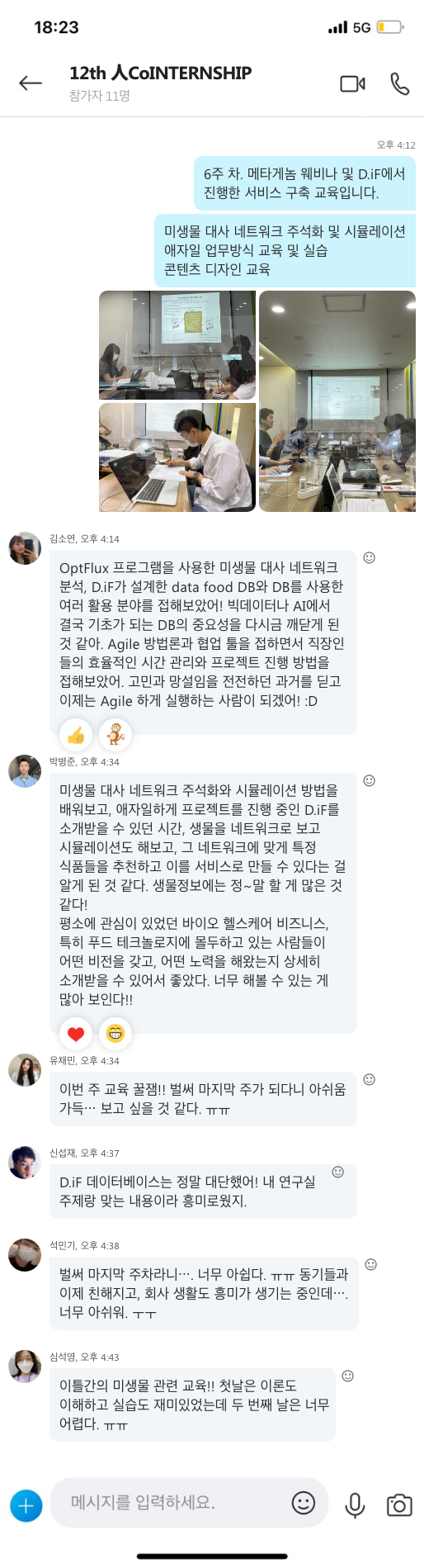
NGS 장비를 활용한 유전체 해독 기술이 지속적으로 발전하면서 이번 COVID-19 사태에서는 메르스 때와는 다르게 감염 환자가 발생하고 늦어도 일주일 안에 유전체 정보가 해독되어 공개되는 것을 볼 수 있습니다.

중국 현장에서는 초소형 유전체 해독 장비인 MinION(Oxford Nanopore)을 활용해 환자 유래 SARS-CoV-2 유전체를 현장에서 24시간 내 시퀀싱 하는 데 활용하고 있으며, 중국 전역에서 발생한 다양한 이 바이러스 유전체 데이터 수백 개가 동시에 계속해서 공개되고 있습니다.

[출처] NANOPORE사의 MinION
MinION을 사용한 시퀀싱은 SARS-CoV-2의 진화를 추적하기 위한 빠르고 효율적인 방법입니다. 실험실 장비에 비용적으로 거의 투자하지 않고 몇 시간 안에 시퀀싱을 수행할 수 있지만, 샘플의 품질이 낮고 다른 출처의 RNA로 오염된 경우가 많기에 생물정보 분석이 굉장히 중요합니다.
이번 블로그에서는 metatranscriptomics 데이터의 한계를 극복하고, CLC Genomics Workbench를 사용하여 Oxford Nanopore MinION 장비에서 시퀀싱된 데이터를 최대한 활용할 수 있는 방법을 안내하도록 하겠습니다.
분석할 데이터는 중국 우한의 해산물 시장에서 처음 폐렴이 발생하였을 때의 샘플을 MinION 장비로 시퀀싱한 메타전사체 케이스입니다[Chan et al., 2020].
환자로부터 가래와 인후에서 면봉을 사용하여 기관지 폐포를 통해 샘플링 하였고, 질병의 직접적인 원인이 되는 바이러스 외의 RNA가 함께 포함되어 있으며, 염기서열에 비의존적으로 무작위 primer를 사용하여 새로운 병원체의 동정이 가능한 SISPA(Single-Primer Amplification) 프로토콜을 사용하였습니다.

CLC Genomics Workbench 20.0 버전 이상에서는 "Long Read Support(Beta)" 플러그인을 지원합니다. 해당 플러그인을 설치하면 Oxford Nanopore의 MinION 장비에서 시퀀싱한 raw 데이터를 import 할 수가 있습니다.
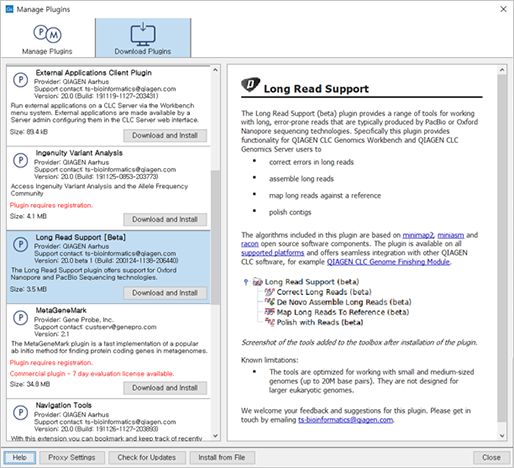
(Long Read Support Plug-in)
직접 시퀀싱한 데이터 외에 공개된 데이터를 기반으로 분석을 진행해보고 싶다면, SRA 데이터베이스를 연결하여 NGS raw 데이터와 메타데이터를 다운로드하여 분석에 사용할 수 있습니다.
분석에 활용한 데이터도 해당 논문 정보를 토대로 가래과 인후에서 면봉으로 채취한 2개의 샘플을 CLC Genomics Workbench내에 연동된 SRA에서 다운로드 해보았습니다.
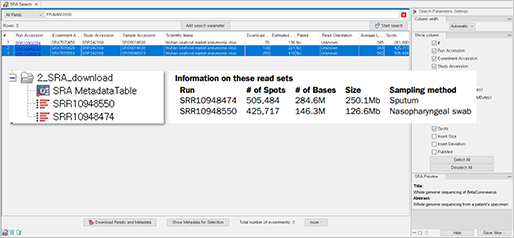
(Search for reads in SRA)
더불어 변이 분석을 위한 참조 유전체 서열도 우한에서 등록된 최초의 SARS-CoV-2 바이러스 서열(MT135044)을 NCBI search 기능을 이용하여 준비하였습니다.
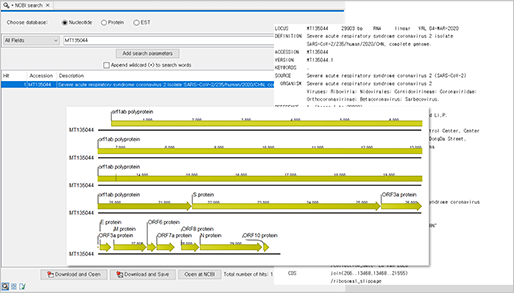
(NCBI search & Download)

해당 샘플에서는 바이러스 서열 농축을 위한 SISPA 프로토콜을 사용하였기 때문에, SISPA adapter를 제거하기 위한 adapter 서열을 별도로 저장한 후 trimming 과정을 통해 시퀀싱된 서열 내에 포함된 adapter를 제거하였습니다.
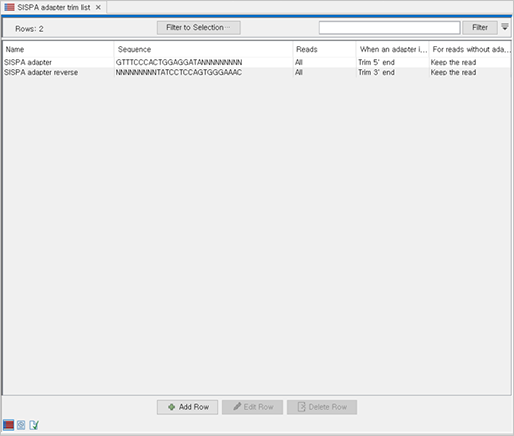
(Adapter trim list)

Long Read Support 플러그인은 PacBio 또는 Oxford Nanopore 장비에서 생산된 long read의 de novo assembly와 reference mapping 분석을 지원합니다.
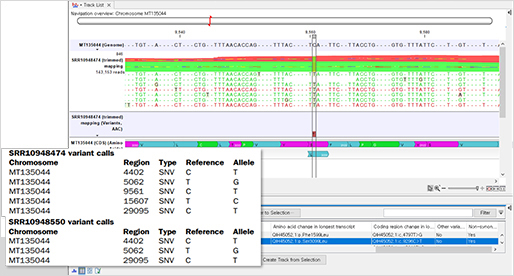
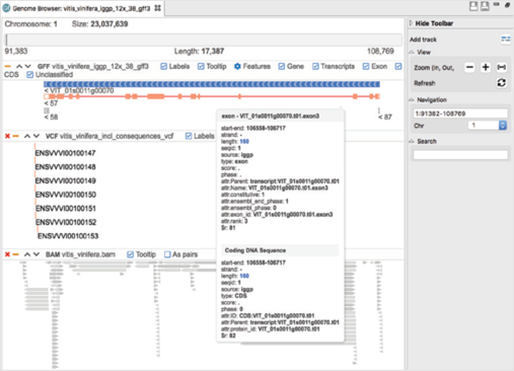
Quality 및 adapter trimming 과정을 거친 2개의 시퀀싱 데이터는 중국 우한에서 등록된 SARS-CoV-2 참조 유전체를 기준으로 맵핑을 진행하였고, 전체 시퀀싱된 서열 중에서 샘플 내 코로나 바이러스 서열이 포함되어 있음을 확인할 수 있었습니다.
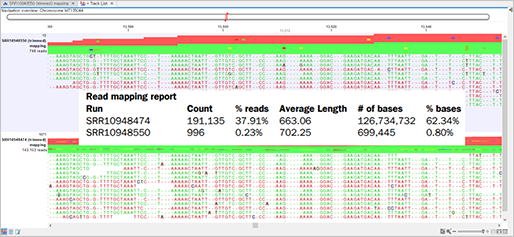
(Read mapping)
맵핑 리포트를 확인해보면, 인후 샘플에서는 채취된 샘플 내에 바이러스가 극소량이라 host genome 데이터가 많이 포함되었을 가능성을 나타냅니다.

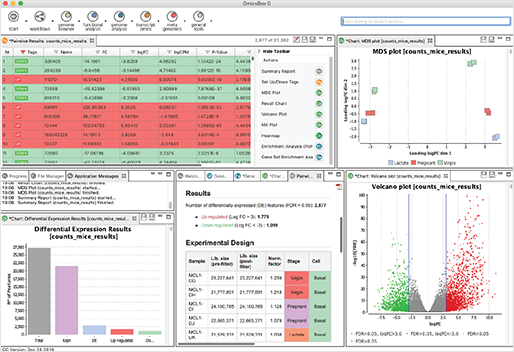
참조 유전체 서열에 맵핑된 리드들을 기반으로 변이 분석을 수행할 때 CLC Genomics Workbench에서는 3가지 검출 알고리즘을 제공합니다.
이 중 Fixed Ploidy Variant Detection의 경우는 유전체 배수체를 기준으로 germline 변이 분석에 최적화된 알고리즘이지만, 시퀀싱 에러 모델도 함께 적용되어 있으므로 Oxford Nanopore 데이터의 변이 분석에 효과적일 수 있습니다.
분석 결과, 가래 샘플에서는 5개의 변이가 발견되었고, 인후 샘플에서는 3개의 변이가 발견되었습니다.
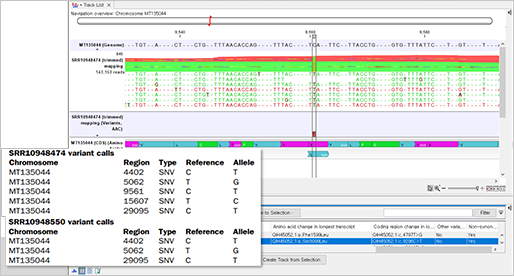
(Variant calls)
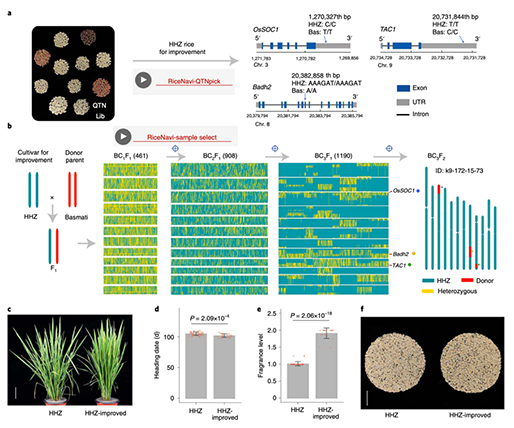
샘플링 날짜를 살펴보았을 때, 참조 유전체 서열인 MT135044의 경우는 2020년 1월 28일이고 MinION 2개의 시퀀싱 샘플은 2020년 1월 11일로 활동적인 발병 상황에서 시간이 지남에 따라 돌연변이를 추적할 수 있음을 나타냅니다.
추가로 해당 변이의 아미노산 변화에 영향을 미치는지도 확인할 수 있으며, 이를 기반으로 단백질 구조에도 변화를 일으키는지 3차원 형태로 확인할 수 있습니다.
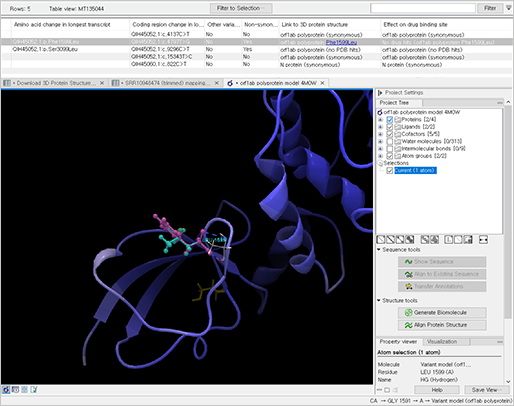
(Protein model of a non-synonymous variant)

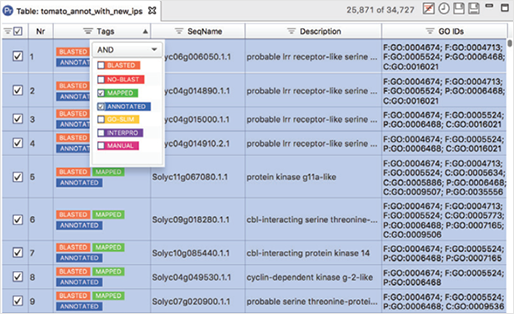
각 샘플별로 참조 유전체 서열에 맵핑된 consensus 서열을 별도로 저장하여 NCBI의 BLAST를 수행하면, 다른 샘플의 SARS-CoV-2 서열들과도 비교할 수 있습니다.
COVID-19 사태로 별도의 BLAST DB도 운영하고 있으므로, 필요 시 local 환경에 Betacoronavirus DB를 다운로드 받아서 Local BLAST 분석도 가능합니다.
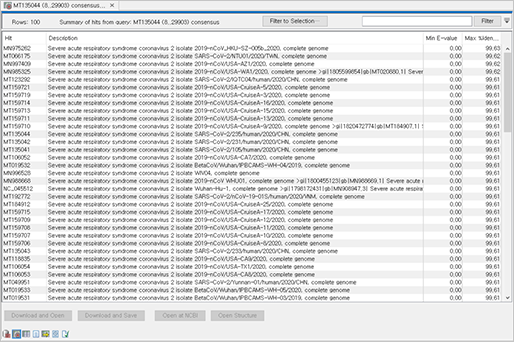
(BLAST output hit table)

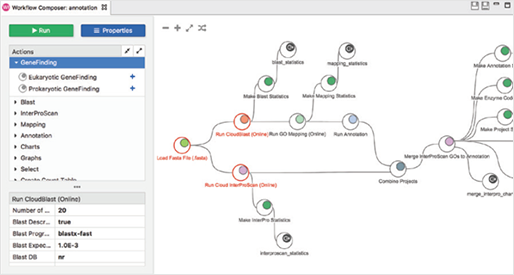
더불어 여기까지 분석은 워크플로우를 만들면 다량의 샘플을 분석할 때 굉장히 효율적으로 쉽게 운영할 수 있습니다.
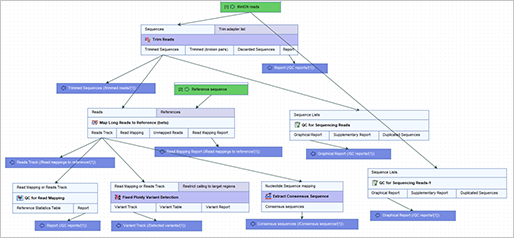
(A workflow reproducing the bioinformatics pipeline)
이처럼 생물정보 분석은 전체 유전체 수준에서 전염병의 원인이 되는 병원체를 분석하고 비교할 수 있습니다. 더불어 메타 데이터를 이용하여 환자 유래의 바이러스 및 병원체의 유전체 정보를 함께 비교하면 발생 원인을 역으로 추적할 수 있습니다.
현재 (주)인실리코젠에서는 COVID-19 연구를 포함한 감염병 연구를 위해 CLC Genomics ProSuite(CLC Genomics Workbench를 포함한 바이러스 및 미생물 NGS 분석 패키지)와 Ingenuity Pathway Analysis(IPA, 유전자 네트워크 분석 솔루션)의 단기 라이선스를 지원하고 있으니 신청하시면 6월 15일까지 사용해보실 수 있습니다.
하루 속히 COVID-19 감염 확산이 종식되길 바라며, 앞으로 이러한 생물정보 솔루션들의 활용을 통해서 바이러스 및 병원체 발생을 조기에 감지하고 제어하는 공중 보건의 보호에 도움이 되길 바랍니다.

- Chan, J. et al., A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. Lancet, 395(15):514-523
- Aiping Wu, Yousong Peng, et al., Genome Composition and Divergence of the Novel Coronavirus (2019 nCoV) Originating in China. Cell Host and Microbe, 27
- GISAID, https ://www.gisaid.org/epiflu applications/next betacov app/
작성 : insilico Lab 김경윤 책임
Posted by 人Co
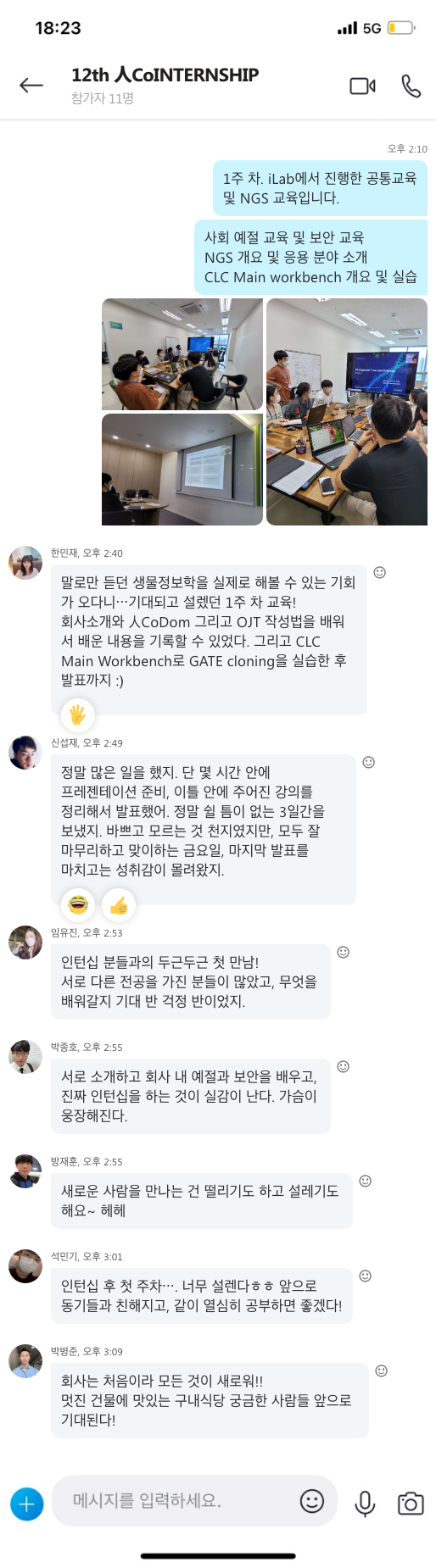
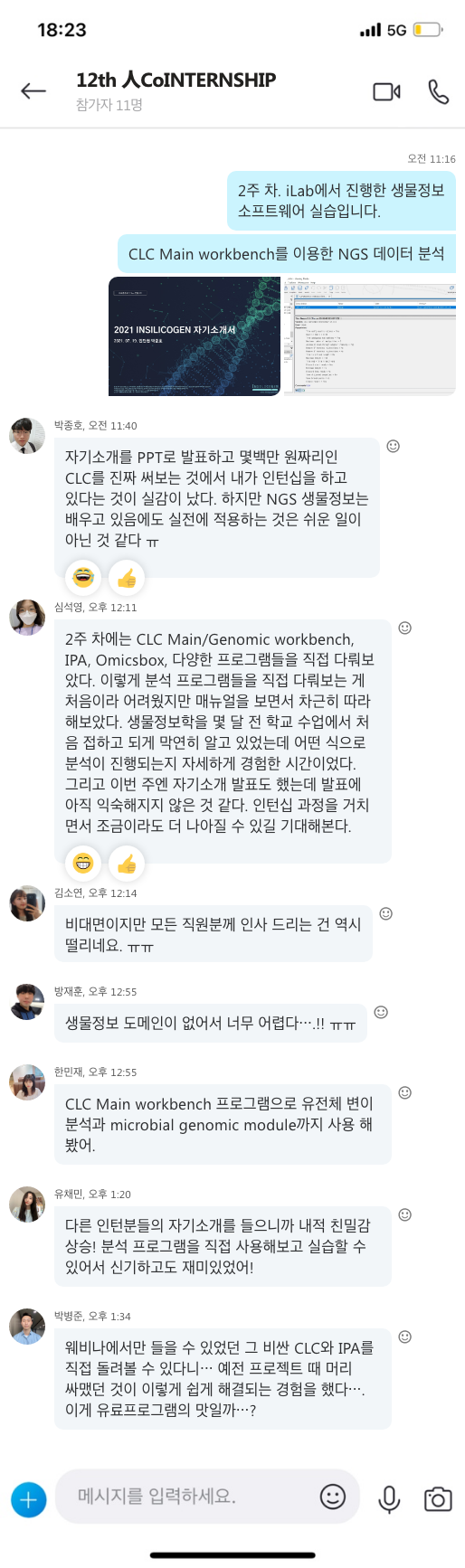
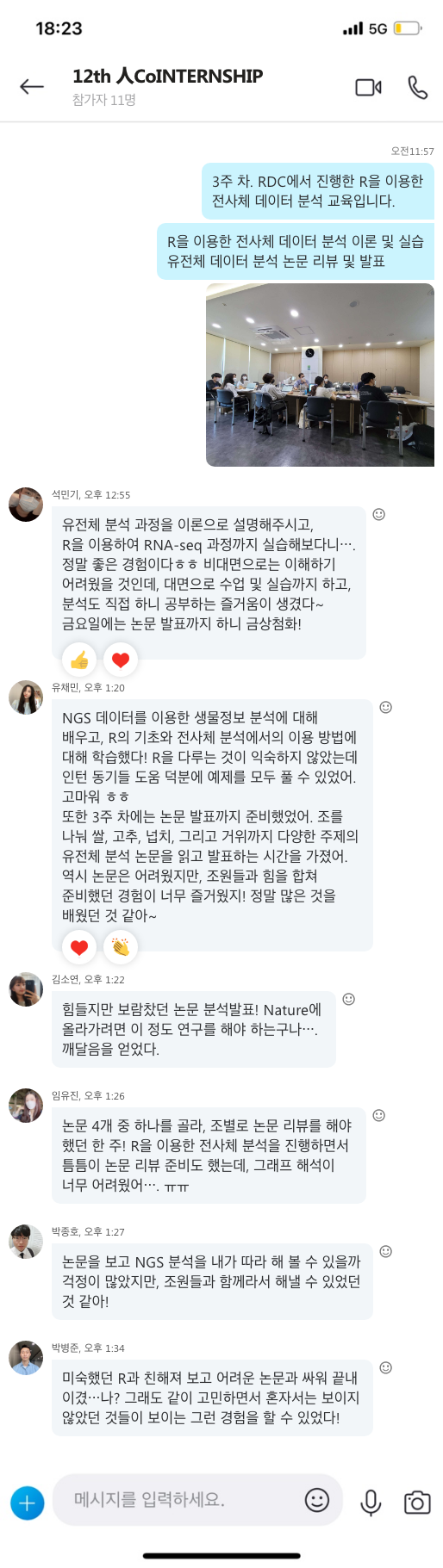


























 [Fig. 1] 전주한정식
[Fig. 1] 전주한정식