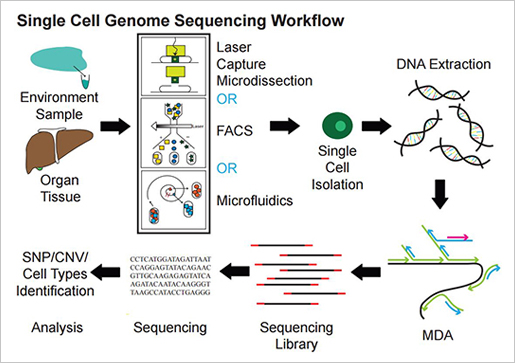
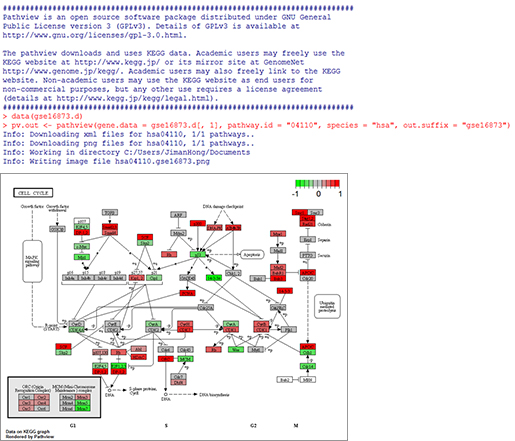
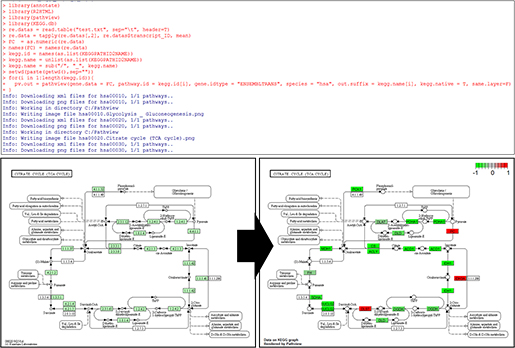
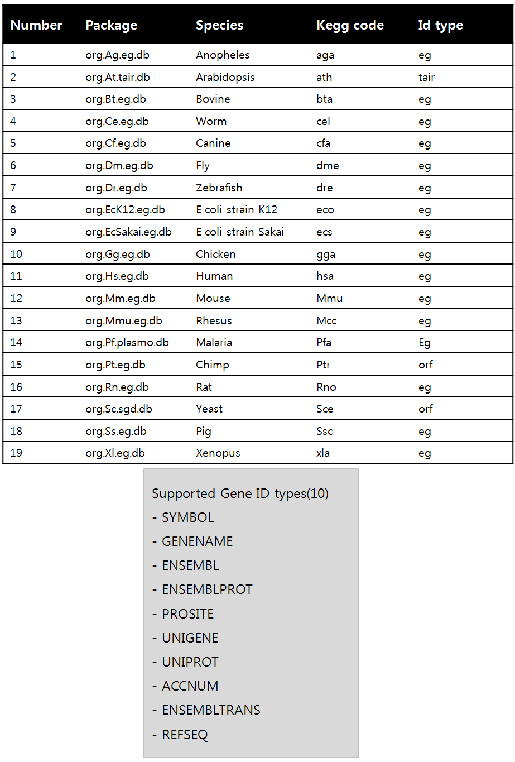
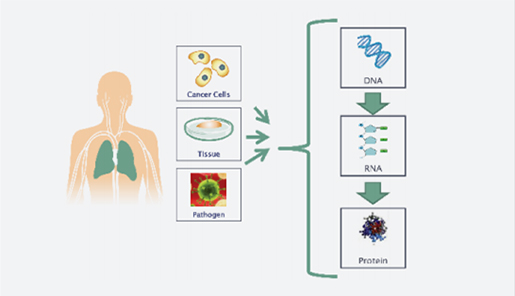
글로벌 바이오의 중심인 생물정보 데이터베이스에서 바이오 융합기술의 미래를 디자인하다
- Posted at 2016/04/19 17:00
- Filed under 생물정보
Qiagen은 안정적인 전장유전체 분석(Whole Genome Analysis) 서비스를 위해 Intel과 제휴를 하였다.
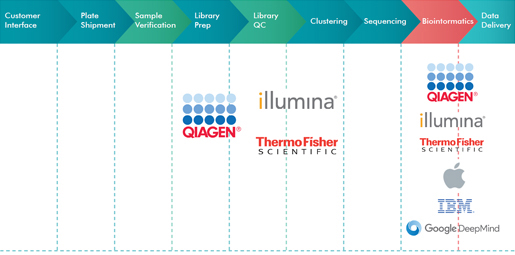


본 솔루션의 유전체 해독 파이프라인은 NGS 분야 과학자들이 효율적인 비용으로 생산성을 증대할 수 있도록 한층 더 자연스럽게 디자인 되었다. 전장 유전체 분석을 진행하는데 발생하는 비용은 22달러(한화 약 25,000원)만으로 가능하게 되었다.
이것은 일루미나 HiSeq X ten이 요구하는 컴퓨팅 성능과 분석의 수요를 만족시킬 수 있다. HiSeq X ten이 요구하는 컴퓨팅 파워는 85-node 클러스터이나, Intel의 혁신적인 기술력이 담긴 32-node 클러스터(28-core E5-2697 v3 @ 2.60GHz, 128 GB RAM)를 통한 BWA + GATK variant calling 파이프 라인은 연구자들로 하여금 굳이 85 node 클러스터를 구입하지 않아도 안정적인 전장 유전체 분석이 가능하도록 하여 1.3 백만 달러(한화 15억 원)를 절약할 수 있도록 하였다.
Qiagen 솔루션의 달라진 점은 아래와 같다.
- 서버에 설치된 분석 도구 : 이 시스템은 Biomedical Genomics Server 솔루션 사용
- 안정성 : 연구실의 컴퓨팅 성능, 네트워킹, 저장용량에 대한 요구에 적합하게 디자인 되어서 분석 용량에 대한 관리가 상당히 비용 효율적이고, 편리한 관리
- 신뢰할 수 있는 정확성 : NGS data 분석에 있어서 분석 효율과 비용 효율이 중요하지만, 본 솔루션은 Variant calling과 해석에 있어서 신뢰할 수 있음
- 사용자 편리성 : 본 솔루션은 클러스터 계산과 같은 복잡한 것들을 마스킹하여 Biomedical Genomics Workbench(BxWB)로 사용하기 편리함
- 신속한 데이터 연결성 : 대역폭이 포트당 최대 40 Gbps Intel True Scale Fabric 기반의 고성능 상호 연결 시스템으로 컴퓨팅 노드와 중앙 집중형 저장 공간과의 연결이 가능함
-
병렬 저장 능력 : 본 솔루션은 모든 컴퓨터의 노드(Node)와 코어(Core) 및 쓰레드(Thread)를 유지하면서도 높은 효율성을 띄는 병렬 저장 시스템인 Intel 엔터프라이즈 에디션으로 통합되었음
Intel의 혁신적 기술력이 담긴 32개 노드 시스템은 평균적으로 24시간 동안 일루미나 HiSeq X Ten이 생산 할 수 있는 48개의 genome을 생산하고 분석할 수 있다. 또한 24시간 안에 대략 1,440 인간 엑솜(Exome) 데이터를 분석 할 수 있다. 2016년 4월 Bio-IT World Conference에서 Qiagen과 Intel의 전략적 제휴를 통한 공동 솔루션을 발표하였다.
전장 유전체 분석에 고성능 CPU를 제공하는 인텔(Integrated Electronics) 은 세계에서 가장 큰 반도체 제조사로 본사는 캘리포니아 주 산타클라라에 있으며, 반도체 제조 공장은 같은 주의 새너제이에 있다. 1968년 7월 8일 고든 무어와 로버트 노이스가 인텔을 설립하였고, 1971년 최초 마이크로프로세서 인텔 4004를 만들었다. 1.5년마다 CPU 속도가 2배가 된다는 무어의 법칙도 인텔의 설립자인 고든 무어가 발표한 내용이다. 인텔은 반도체 뿐 아니라 메인보드 칩셋, 네트워크 카드, 집적회로, 플래시 메모리, 그래픽 프로세서 및 임베디드 프로세서 등 통신과 컴퓨팅에 관련된 장치를 만들면서 그 사업의 영역을 무한히 확장하였다. 2012년부터 지속적으로 매년 52,708 백만달러(한화 60조 원)이상의 매출을 내고 있으며, 2014년 64조 원이라는 사상 최대 매출액을 기록하였고 2015년은 63조원으로 전년대비 약 1% 감소하였다.
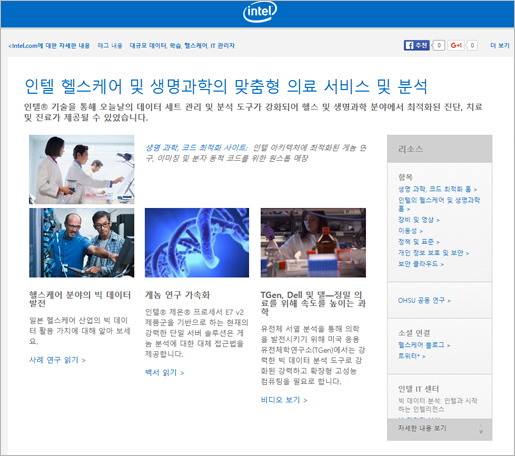
컴퓨터의 가장 핵심인 인텔 CPU의 급속한 발전은 유전체 해독기술의 발전 및 유전체 해독비용 감소에 큰 영향을 미쳤다. 앞서 열거한 IT 회사들과 같이 인텔도 기존의 IT 시장에서 벌어들인 수익으로 또 다른 미래 먹거리 발굴에 열정을 보이고 있으며, 그 중 스마트 헬스케어를 위한 IoT 기술 활용에 투자를 아끼지 않고 있다. 인텔은 2007년 헬스케어 부문을 성장 동력 중 하나로 보고 생명과학 부분에 투자하였고, 2015년 8월 암환자의 유전자 및 임상 데이터 공유가 가능한 클라우드 플랫폼인 CCC(Collaborative Cancer Cloud)를 발표했다. 이 시스템은 병원 및 연구기관에서 환자의 유전자 정보, 의학용 시각 자료, 임상 데이터 등을 공유한다. 또한 헬스케어 시장에 최적화된 제온(Xeon) CPU를 개발하였고, CareWork 라인을 만들어 워크스테이션 및 서버 제품을 전세계 각 의료기관에 공급하고 있다.
(주)인실리코젠은 다양한 생물학적 데이터 분석을 효과적으로 공유하고 소통할 수 있는 플랫폼을 제공하고 있다. Bionformatics, Deep in Big 이라는 기치아래 고객과 함께 정보의 축적을 넘어 새로운 가치 창조를 실현하고자 한다. 이를 위해 11년이라는 기간 동안 생물정보학이라는 한 길을 걸어오며 그 가치를 실현해 왔다.
필자는 대한민국에 생물정보학이 단순히 학문 또는 기초 산업인 시퀀싱에서만 그치지 않고 생물정보 산업 및 데이터베이스로 발전할 수 있었던 것은 많은 부분 (주)인실리코젠의 끊임없는 노력 때문이라고 본다. 본 기업의 지속적인 노력으로 인해 각 정부 기관에 다양한 생물 관련 데이터베이스가 구축이 되었고, 각 연구기관 및 산업현장에 고도의 생물정보 분석을 위한 시스템, 소프트웨어 및 분석 컨설팅이 제공 되어왔다. 이로 인해 생물정보라는 산업은 기초 산업을 넘어선 고급 산업의 영역으로 도약할 수 있었고, 이를 기반으로 다양한 기술과 융합을 통한 새로운 미래를 그릴 수 있는 BI 플랫폼을 창조할 수 있게 되었다. 생물정보 산업의 발전을 위해 (주)인실리코젠이 이뤄낸 결실은 실로 대한민국 생물정보의 산실이었다고 평가할 만하다.
이지현 주임
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/206









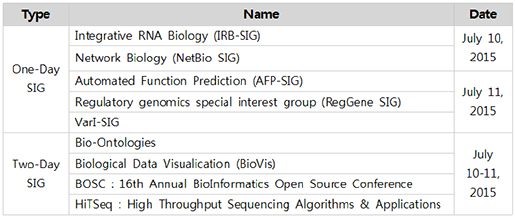
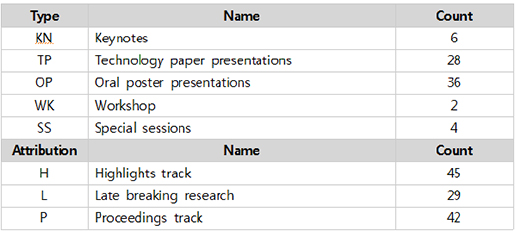


 Figure 2. Exhibitors of ISMB/ECCB2015
Figure 2. Exhibitors of ISMB/ECCB2015