TCGA 유방암 WES 데이터로 CNA 탐지도구 정확도 확인 - Oncotarget 논문 게재
- Posted at 2017/05/30 08:44
- Filed under 생물정보
암을 유발하는 유전변이 가운데 가장 중요한 것이 복제수변이(Copy-number variation, CNV)입니다. 암 복제수변이는 체세포(somatic) 유전변이이기 때문에 생식세포 복제수변이(germline CNV)와 구분하여 CNA(Copy-number alteration)라고도 합니다. 유전체의 특정 유전자 영역이 증폭(amplification)되거나, 삭제(deletion)됨으로써, 온코진(oncogene) 강화 혹은 종양억제유전자 약화 역할을 수행합니다. 치환변이(SNV)도 중요하지만, 그 종류가 너무 다양하기 때문에 치료 표적으로 삼기가 복잡하지만, CNA는 해당 유전자를 직접 억제하거나 보완하도록 치료표적으로 할 수 있기 때문에 임상에서 더욱 중요합니다.
특정 암 조직에 대해 유전체 복제수변이 CNA가 있는지 확인하는 다양한 방법이 있습니다. 고전적인 FISH 등 염색 후 현미경으로 관찰 방법에서, 고밀도 SNP array의 방법으로 발전해 왔고, 특히 SNP6라고 알려진 Affymetrix의 칩은 SNV과 함께 CNA를 탐지하는데 널리 사용되고 있습니다. 최근 NGS 실험방법의 발전으로, WGS, WES 데이터로 매핑정도(mapping depth)를 이용하여 CNA를 추정할 수 있는데, 이는 정밀의료시대를 위해 중요한 분석 방법으로 주목 받고 있습니다. NGS 데이터로 SNV와 CNA를 함께 탐지하고, 유전변이에 맞는 치료를 수행할 수 있기 때문입니다.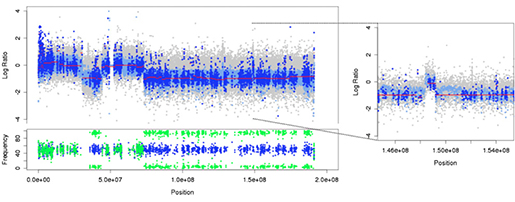
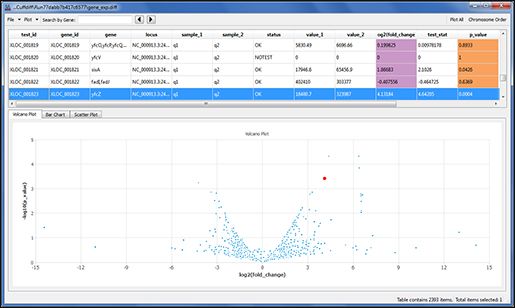
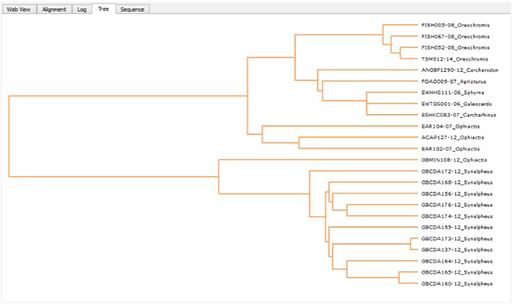
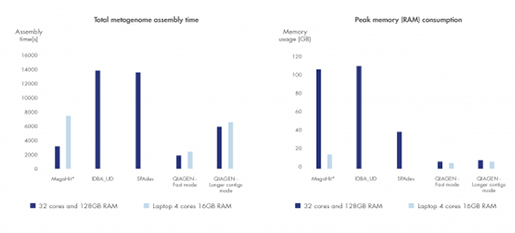
(그림 1. VarScan2 프로그램이 WES 데이터로 mapping depth를 기반으로 CNA 추정하는 과정 - 염색체의 특정 영역이 삭제되거나 증폭됨을 알 수 있습니다. 출처: Exome-based Copy Number Analysis with VarScan2)
다양한 프로그램들이 NGS 데이터로부터 CNA를 탐지할 수 있습니다. 보통은 BAM 파일을 읽어 유전체의 어느 영역이 CNA인지 추정합니다. 다양한 알고리즘들이 사용되지만, 각각의 특징들로 인해 그 정확성은 다양합니다. NGS 기반 정밀의료를 위해서는 어떤 방법이 정확하게 NGS 데이터로 CNA 추정할 수 있는지 확인하는 것이 중요합니다.
한양의대 공구 교수님 지도로 TCGA 유방암 WES 데이터로 7종의 WES CNA 탐지도구의 정확도를 평가한 연구 결과가 Oncotarget에 실렸습니다. (Gene-based comparative analysis of tools for estimating copy number alterations using whole-exome sequencing data Oncotarget 2017)
TCGA는 암 환자의 WES 데이터 뿐 아니라, SNP6로 실험한 CNA 데이터를 함께 제공합니다. 이번 연구는 TCGA에서 제공되는 SNP6 CNA 데이터를 정답으로 하여, 다양한 WES 기반 CNA 탐지 프로그램(CoNIFER, CODEX, ngCGH, ExomeCNV, VarScan2, saasCNV, falcon)의 정확도를 확인하였습니다.
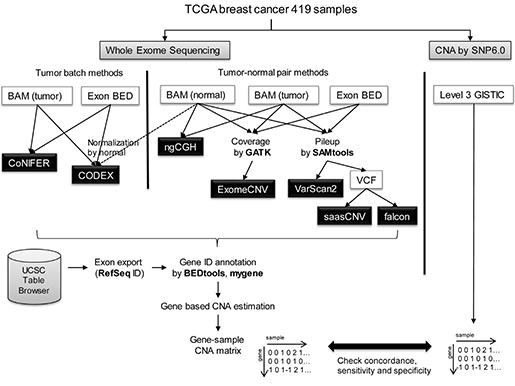
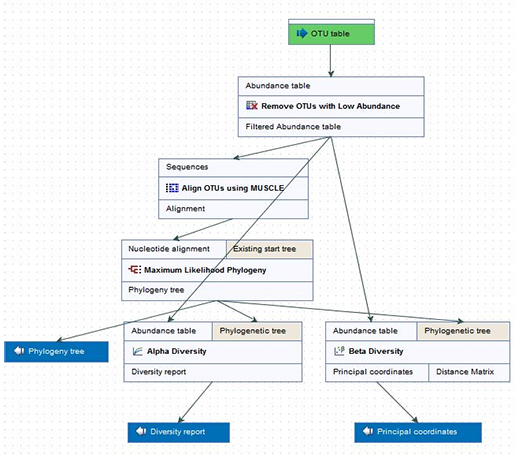
(그림 2. 본 연구방법의 전체 모식도 - TCGA 유방암 419 사례의 WES CNA 추정결과와 SNP6 CNA 결과를 비교함)
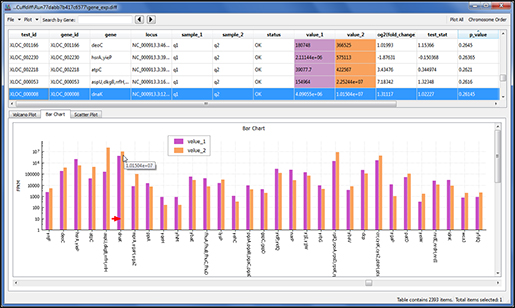
TCGA 유방암 419 사례에서 각각 민감도(sensitivity)와 특이도(specificity)를 확인한 결과는 다음과 같습니다.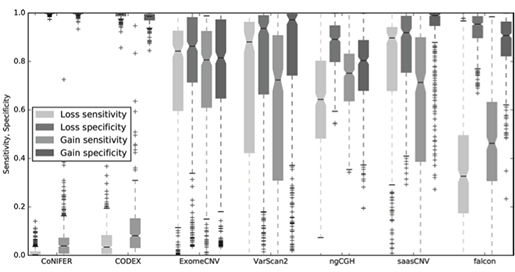
(그림 3. 7개 CNA 추정 프로그램의 민감도, 특이도 막대그래프. CNA Gain과 Loss로 나누어 각각 확인함)
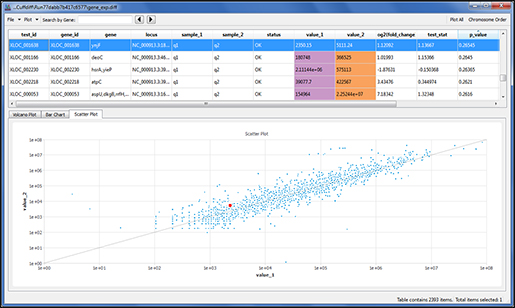
하나의 사례를 골라서 프로그램마다 얼마나 결과가 유사한지 확인한 결과는 다음과 같습니다.
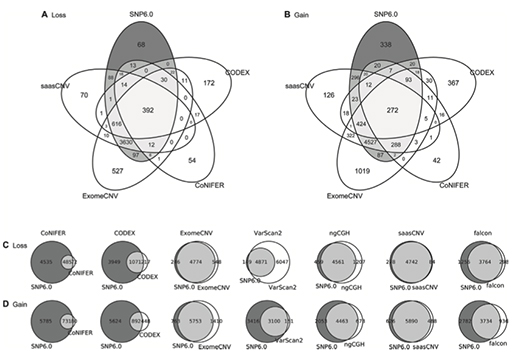
(그림 4. 하나의 사례에서 CNA Gain/Loss를 정답인 SNP6 결과와 비교하고, 그 결과를 벤 다이어그램으로 표시)
전반적으로 암-정상 사례로 분석하는 도구(ExomeCNV 등)가 암 사례만 분석하는 도구(CoNIFER 등)에 비해 정확도가 높았습니다. 본 연구를 통해 CNA를 정확하게 추정하기 위해서는 정상조직도 함께 NGS 분석해야 함을 확인할 수 있었습니다. 종합적으로 saasCNV 프로그램이 가장 정확도가 높았습니다. 이 프로그램은 복제수를 대립유전자별로 확인(allele specific CNV caiing)할 수 있는 장점도 있어서 앞으로 NGS 데이터로 CNA를 추정하는데 중요하게 활용될 수 있을 것으로 기대합니다. 또한, 어떤 사례는 정확도가 높고, 어떤 사례는 정확도가 낮은데, 샘플 데이터의 어떤 요인이 정확도에 영향을 미치는지도 추가로 연구하여, 정밀의료 진단을 위한 분석 방법으로 활용 할 수 있습니다.
본 연구를 수행하는데 가장 많은 도움을 준 것은 Jupyter와 pandas입니다. "419사례 x 2만여 유전자" 행렬을 다양하게 다뤄야 하는데, pandas로 어렵지 않게 할 수 있었고, 중간중간 분석 결과들을 jupyter로 관리할 수 있었습니다. 이들 도구를 잘 사용하는 것은 유사한 분석을 수행하는데 필수 불가결한 요소가 될 것입니다.
작성자 : Platform Lab 수석개발자 김형용
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/246


 입사지원서_성명_지원부문2017_20170517.docx
입사지원서_성명_지원부문2017_20170517.docx