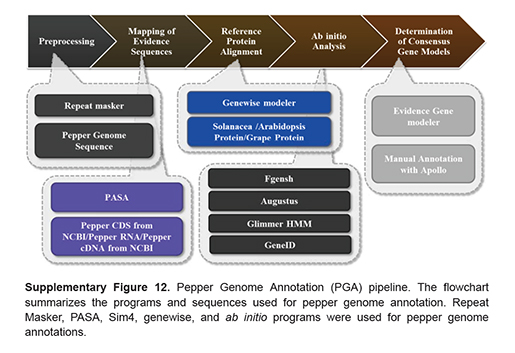
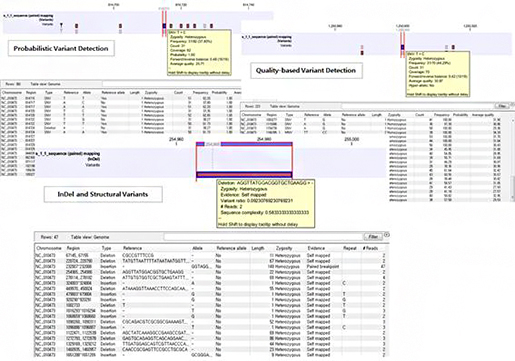
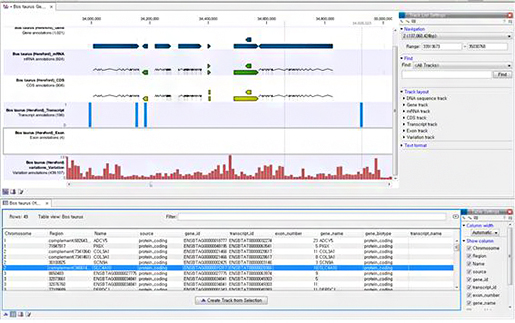
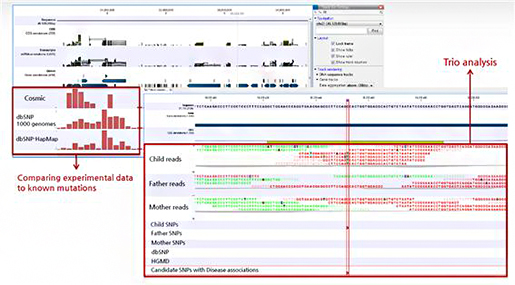
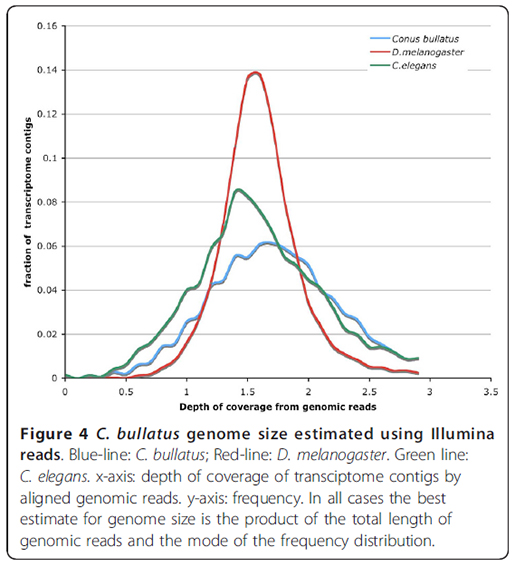
지난 1월 7일부터 한달여간 (주)인실리코젠의 생물정보 인재양성 프로그램인
人Co
INTERNSHIP 프로그램이 진행되었습니다. 국내 기업과 연구기관에서 요구하는 최신의 생물정보 솔루션과 기초 기술교육 및 사내
현장실습을 통해 바이오 연구개발과 조직 생활의 기초 개념을 이해할 수 있는 좋은 기회를 제공하는데 목적을 둔 본 프로그램에
앞으로도 생물정보인재로 성장하고자 하는 많은 분들의 관심과 참여 바랍니다.
수료하신 네 분 모두 축하드리며, 소감을 한 번 들어볼까요?
강전모
2013 人Co Internship에 참여한 강전모입니다. 전 이번 2013년 새해를 인실리코젠과 함께 시작하였습니다.
지금 영남대학교 생명공학과에 재학 중에 있지만 생명공학
전공임에도 불구하고 전공 관련 기업 등에 대해서는 잘 알고 있지 않았습니다. 그러던 중 동기생인 명희선배의 취업소식을 통해
'인실리코젠' 이라는 회사를 처음 알게 되었고, 이 후, 학교에서 열리는 학회에서 박준형 부장님의 발표도 보고, 강의시간에 정명희
선배의 회사 소개를 들으면서 인실리코젠에 대해서 알게 되었습니다.
이렇게 알게 된 인실리코젠에 대한 첫 이미지는 빼어난
디자인이었습니다. 아직 잘 알지 못했었지만 다른 곳과 다르게 딱딱하지 않은 홈페이지와 블로그는 부담 없이 알아갈 수 있도록
도와주었습니다. 호감을 가지고 둘러보던 중 인실리코젠의 기업가치인 人Co ‘사람(人)을 중심(Core)으로
컴퓨터(Computer)를 통해 배려(Consideration)하고 소통(Communication)한다.’ 를 보고 감동
받았었습니다. 또 제가 관심이 있는 컴퓨터를 이용하고, 사람이 중심이라는 말을 통해 호감은 관심으로 이어졌습니다. 이 관심은
저에게 경험을 위해 한 발짝 내딛을 수 있는 용기를 주었고 이를 계기로 이번 12월 학교 홈페이지를 통해 알게 된 ‘인턴십 지원자
모집’ 과 교수님의 추천으로 이번 인턴십에 지원하여 참여할 수 있게 되었습니다.
1월7일 떨리는 마음으로 인실리코젠에 출근한 첫날, 자리와 노트북을 배정받고 회사 업무에 필요한 skype나 wiki 사용법, 회사
소개 등을 받으며 인턴십을 시작하였습니다. 무엇보다 송하나 선배가 직원 한분 한분을 소개시켜주시는데 반갑게 맞이해주시며 정중하게
명함을 건네주시던 그 모습이 정말 인상 깊었습니다. 첫 주의 수요세미나를 통해 각자 자기소개 발표를 하며 정식으로 직원 분들께
인사를 드렸는데, 발표 전 까지는 괜찮았지만 발표를 시작하는 순간 너무 떨리는 바람에 어떻게 발표했는지 기억도 안날 정도였습니다.
1주차 중반에 접어들면서 CLC Main
Workbench에 대해 배우기 시작했습니다. 수요세미나 후 농림수산검역검사본부에 CLC Main workbench 강의지원을
나가서 책자와 볼펜, 설문지도 나눠드리고, 쉬는 시간을 위한 다과도 준비하며 강의 준비를 도와드렸고, 앞으로 익혀나가야 할 CLC
Main Workbench에 대한 강의도 들었습니다. 생각 외로 많은 분들이 모이셨고, 단상에 서서 강의하는 김경윤 주임님과
송하나 선배를 보면서 왠지 모를 뿌듯함과 자신감이 생기는 것 같았습니다. 강의를 들으면서 대략적인 CLC Main
Workbench 사용법을 배웠고 이를 바탕으로 CLC Main Workbench overview를 발표 자료를 만들면서
tool사용법을 숙지하였습니다. overview발표 후 CLC Main Workbench를 이용해 당뇨병과 인플루엔자에 관한 두
가지 case study를 실습하였습니다.
당뇨병에 관한 주제는 '사람의 당뇨병에 연관된 유전적 변이 확인'으로, 사람과 유사한 점이 많은 돼지의 유전체내 당뇨병 관련
위치에서의 서열 다형성을 CLC Main Workbench를 사용하여 분석하는 것이었습니다. CLC Main Workbench에
있는 NCBI 검색기능으로 당뇨병관련 유전자인 UCP유전자를 찾고, 이 유전자를 BLAST하여 상동성을 가진 돼지 및 다른 개체
5종의 서열을 다운받아 Alignment를 수행한 후 나머지 종들과 비교하면서 돼지와 사람에서 SNP로 의심되는 지역을
annotation 하였습니다. 이 후, NCBI SNP DB에 올라와있는 SNP를 비교해 실제 SNP가 존재하는 지역을 찾고
이를 포함하는 유전자 서열을 클로닝하기 위한 primer도 제작해보고, 임의로 선택한 증폭구간의 서열에서 SNP가 바뀌어서
아미노산 서열이 바뀌는 지를 translation tool을 이용해 비교해보았습니다. 두번째는 인플루엔자에 관해 '높은 발병률의
조류독감 바이러스의 유전적 특성'을 주제로 case study를 진행하였고 당뇨병의 case study와 비슷한 방법으로
수행하였습니다. CLC Main Workbench를 이용한 DB search를 통해 각 type 별 인플루엔자 서열을 받아
Alignment를 수행하고, 이를 통해 type간의 서열차이를 확인하고 BLAST를 이용해 각 type별 인플루엔자의 상동성을
조사하였습니다. 이 과정에서 각 인플루엔자 간에 동일하게 일치하는 지역을 확인하였고, 이는 인플루엔자 연구에서 primer 제작
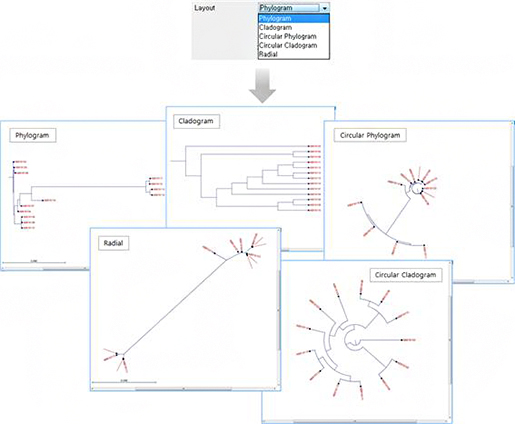
등에 사용될 수 있을 것이라는 생각도 하게 되었습니다. 마지막으로는 phylogenetic tree 그려 봄으로 인플루엔자
바이러스가 지역별로 분화한다는 특징도 확인하였습니다. 이 두 case study에 대한 발표준비를 하면서 인턴십에 참여하지
않았다면 알 수 없는 CLC Main Wrokbench라는 프로그램을 알게 되었고, CLC Main Workbench가 실험이나
연구에 얼마나 많은 도움을 줄 수 있는지도 몸소 체험하는 계기가 되었습니다.
이후, Genomics Workbenh에 대해 김경윤 주임님의 설명을 듣고 Main Workbench처럼 그룹스터디가 아니라 개별
스터디로 발표 주제를 각각 배정받고 실습했습니다. 학교에서 수업으로만 들었던 NGS 데이터를 이용해 assembly를 실습할 수
있었는데, assembly에 관한 알고리즘이나 각 NGS file format에 맞는 옵션설정과 assembly를 수행하기 위한
parameter설정 등의 절차를 알게 되면서 학교에서는 미처 배우지 못했던 것을 알게 되는 계기가 되었습니다.
인턴십 시작할 때 책을 한권씩 나눠 주셨습니다. '사원의 마음가짐'이라는 책이었는데 회사에 실습하면서 책을 읽게 될 줄은 예상
밖이었습니다. 예전엔 서점에서 책도 구매하여 읽고, 좋아하는 작가도 있었으며 책 읽는 것을 좋아했는데 지금은 거의 읽어본 적이
없어서 내심 반성하는 기회도 되었습니다. 이 책을 읽고 난 후, 지금 가장 크게 와 닿았던 문구가 있습니다. '백문이 불여일험'
'백번 듣는 것 보다 한번 경험하는 것이 더 좋다.' 라는 뜻입니다. 4주 동안 CLC Main Workbench와
Genomics Workbench를 배우고 실습하면서 사실 종종 의문점도 들었었습니다. 인턴십 과정 이기는 하지만 회사의 업무를
배워 간단한 일이나마 도와드린다는 생각보단 회사에게 일방적으로 배우기만 하는 과정이 더 컸기 때문입니다. 하지만 점점 발표준비를
하고 사장님과 이사님 그리고 부장님, 주임님의 면담과 직원 분들이 일하시는 모습을 보고 그냥 지나가는 인턴십 과정이 아니라는 것을
깨달았습니다. 더 좋은 솔루션을 만들고, 좋은 프로그램을 다른 사람들에게 제공하기 위해서는 자신도 프로그램에 대해서 잘 알고
있어야하며, 사용하면서 생긴 여러 가지 문제점들과 의문점을 해결해 줄 수 있어야 하기 때문입니다. 또한 이러한 과정을 통해 자신
스스로가 성장하고 이 성장이 회사가 성장하는 계기가 되는 것처럼 하루하루가 그냥 지나가는 것이 아니라 경험이고, 이 경험들이
쌓이면 능력이 된다는 것을 최근에서야 깨달았습니다. 이 뜻을 이해했을 때 인턴십 프로그램에 참여하길 정말 잘했다는 생각이
들었습니다.
또
이번 인턴십에 참여하게 되어 저의 시야를 조금 더 넓힐 수 있게 된 것 같습니다. 사실 생물정보학 분야가 이렇게 넓고 다양할
줄은 생각하지 못했었습니다. 생물정보분석과 실험은 별개라고 구분했던 편견이 깨어졌고 물론 많은 고민이 생기긴 했지만, 저에게
부족한 점이 무엇인지, 무엇을 해야 할지 등의 제가 가야할 길의 갈피를 잡아주었습니다. 아직 1년의 시간이 남은 만큼 조금 더
고민하고, 노력한다면 1년 후에는 더욱 성숙해져있는 저를 볼 수 있을 것 같습니다. 4주간의 人Co Internship을 통해
많은 가르침을 주신 사장님 및 직원 분들에게 감사의 인사를 드립니다.
김정민
4주간 인실리코젠과 저와의 인사
지금(2013년 2월 1일)으로부터 약 2달 전 교수님께서 전화가 오셨습니다. “정민아, 이번에 인실리코젠에서 여름때 못갔던 인턴십을 다시 하니까 갈 수 있지?, 가서 많이 배워왔으면 좋겠다.” 라고 하신 것이 이곳 인실리코젠과의 대면을 위한 교수님의 배려이자 저에게는 첫 걸음마였습니다. 그 후 1달 뒤 처음 수원에 올라올 때, 네비게이션의 목적지가 인실리코젠이 있는 벤처밸리여서 일생 처음 수원에 올라와서 첫발을 내딛은 곳도 ‘회사 바로 앞’이었습니다. 그날따라 날이 춥고 먼 길을 오느라 피곤했지만 낯선 이곳이 신기했고 빨리 회사에 들어가서 회사분들을 뵙고 싶다는 생각을 했습니다. 하
지만 어느 곳이나 마찬가지듯이 낯선 곳은 두렵고, 어색하기 마련이고 여기서 처음 또한 그러했지만, 人co core라는 말과 걸맞게
사람을 가장 먼저로 여기고 소중히 생각하는 이곳의 선배님들께서 너무 자상하게 대해주셔서 적응하는데 큰 힘이 들지 않았습니다.
첫
날, 둘째날에 걸쳐 많은 분들께서 명함을 주시며 특히 정은미 이사님께서 “명함을 대할 때 우리는 그 사람을 대하듯이 소중하게
대한다.”라 말씀하시며 그 분들에게 얼굴이 되어주는 인실리코젠 특유의 디자인이 되어있는 명함을 소중히 받는 시간을 가졌습니다.
실제로 모 교수님을 뵐때 명함을 받으시는 모습을 볼 수 있었는데 받으신 이후에 명함을 대하는 모습을 유심히 보았습니다. 그 결과
저는 명함은 그 사람을 대면할 때 절대로 함부로 하지않고 소중히 앞 테이블에 놓아서 이야기를 나누신다고 우리에게 교육하신 그대로
몸소 실천하고 계심을 보고 진심으로 감명을 받았습니다. 다른 곳에서는 명함같이 작은 것 하나까지 소중히 여기시는 분들을 볼 수가
없었는데 그런 모습을 보면서 또 한번 ‘정말 나는 인턴십을 온 것이 아니라 “人Co” 인턴십에 와있는구나.’ 라는 생각을 하게
되었습니다. 임천안 이사님께서 우리에게 보안각서 등을 주시며 그 순간까지도 깨알같은 조언이나 인생에 있어서 필요한 충고를 아끼지 않으셨습니다.
이
곳에서 가장 기억에 남는 것들 중 하나가 발표하는 것들에 대한 역량을 늘리기 위해서 자주 발표를 하는 시간을 가졌는데 그 시작은
인턴쉽 3일차 되는 날에 가졌던 나에 대한 소개 발표였습니다. 그날의 기억이 아직도 생생한데 많이 긴장하고 떨렸지만 발표할 때에
이곳 분들이 편안하게 하라고 편안한 분위기를 자연스럽게 만들어 주신 대한 작은 배려가 저에게는 매우 큰 힘이 되어 무사히 마칠 수
있었습니다.
또한 우리가 3주가량에 걸쳐 배운 CLC main workbench, CLC genomics workbench라는 프로그램을 처음
접할 수 있게 농림수산검역검사본부에서 열린 세미나에 참석했는데 김경윤 주임님, 송하나 선배님께서 발표하시는 모습을 보게 회사에서
하루일정을 배려해 주셨습니다.
첫 주가 이렇게 마무리 되고 두 번째 주는 본격적으로 CLC main workbench를 이용해서 실습하고, 공부하는 시간을 가졌습니다. 처
음에는 매우 낯선 프로그램을 접함으로 인해 오는 스트레스와 압박감이 있었지만 사용을 하면 할수록 너무 편리하고 유익하고 어딜
가든지 회사의 일원은 아니지만 소개해주고 싶다고 생각이 드는 프로그램임을 깨달을 수 있었습니다. CLC main
workbench를 이용하는 작업을 하면서 이곳에서는 직접적으로 하지는 않았지만 실험적인 측면이나, 기타 생물학적인 배경지식을
쌓는데 많은 도움이 되어 유익한 시간임을 또 한번 깨닫고 이곳에 와서 이렇게 교육을 받는다는 자체가 ‘알아가는 행복’임을
느꼈습니다. CLC genomics workbench를
할 때에는 조금 더 어려운 NGS를 이용한 mapping분석이며, 변이에 대해 분석-조사 했는데, 항상 경윤 주임님께서 “어려운
부분이 있으면 언제든지 와서 물어보고 또 부담감 가지지말고 공부하고 준비해서 아는 만큼 발표하자”고 우리를 위해 배려차원에서
말씀해주셔서 마음을 편히 가지고 공부할 때도 더 집중할 수 있게 되었고 발표 후 경윤 주임님의 설명을 통해 공부할 수 있는 질보다
더욱 심도있는 내용을 공부 하였습니다. 단언코 만일 저러한 프로그램이 필요한 실험실이나 회사가 있다면 돈을 주고 직원을 보내는
손해를 감수하면서도 여기서 교육을 통해 배울 충분한 가치가 있는 내용의 인턴십 프로그램이었습니다.
또한 사람을 먼저 생각하고 그 사람의 인성이나 하고자하는 성실감을 먼저 보며 그 사람의 부족한 점은 시간을 들여서 키운다는 신념아래
저희에게 나눠주신 지금은 고인이 되신 마쓰시타 고노스케씨의 사원의 마음가짐이라는 책을 읽으면서 회사의 일부분으로써 어떠한
마음가짐을 가져야하고 이곳에서 원하는가를 알 수 있었습니다.
다른곳의 인턴십을 체험해보지 못해서 잘은 모르겠습니다만, 이곳은 학교에 제출하는 우리의 임무임에도 불구하고 하나부터 열까지 꼼꼼하고
세세하게 다 신경써주셨던 이지혜 주임님과 모든 과정을 관료적이지 않고 ‘내가 정말 나에 대해서 알고 싶어하는 회사에 있구나’라는
생각이 들게끔 해주었던 모든 프로그램들은 이곳에 오고싶다고 하는 분들이나 관심을 가지고 있는 분들께는 지금이라도 당장 가보라
말씀드리고 싶습니다.
제가 다른 곳을 선택 할 수도 있었지만 교수님께서 “이곳은 가 보아야하는 곳이다”, 학교 선배이신 명희 선배님께서도 “나는
우리회사가 너무 만족스럽고 거기의 사람들이 너무 좋아서 일할 맛이 난다.”라는 말들을 듣고 택했는데 아주 탁월한 선택이었던 것
같습니다. 또 이곳에서는 멘토, 멘티라는 것이 있어서
지정해놓은 멘토에게 궁금한거나 힘든일이 있으면 자연스럽게 가서 여쭈어보고 맘을 터놓을 수 있게 해놓아서 너무 좋았습니다. 제
멘토는 Descign팀의 조아영 주임님이셨는데 1:1로 대화하기에는 너무 바빠보이셔서 그렇게 잘 하지는 못했지만 처음에 적응을 할
수 있게끔 교육때 편의도 봐주시고, 식사도 같이하면서 다른 분들보다는 조금 더 가까워 질 수 있게 해주셔서 감사했습니다. 보
통 무슨 일정이든 끝이나고 돌아갈 때는 더 잘할거라는 아쉬움과 끝이나서 홀가분한 마음이 들기 마련인데 4주교육을 마치고 돌아가는
순간에도 한주만 더 있어서 더 많은 것을 배우고 싶은 마음이 간절해서 제가 제 자신에게 놀래면서 대구로 돌아갑니다.
영리를 추구하는 회사에서 실적이나, 자신의 업무에 대한 책임은 어느곳에나 있듯이 이곳에서도 있겠지만 이곳은 ‘돌봄’이 있는 회사이고 배려가 있는 회사입니다. 높은 직급을 가지신 분께서 사원분들이나, 저희에게 작은 것 하나도 시키지 아니하시고 도와줄 수 있느냐고 말씀하시는 것이 이상적으로 말하는 소위 ‘수평적 관계’의 회사의 바람직한 모습을 보여주고 있지 않느냐 느껴짐을 얻었습니다. 이곳을 안 것은 앞으로의 Bioinformatics 분야로 가기를 마음먹은 저에게는 놓쳐서는 안될 큰 기회이자 행운이었던 것 같습니다. 끝
으로 우리 곁에서 있는 듯 없는 듯 지켜보아주신 최남우 사장님께서는 모든 직원분들의 으뜸되시는 CEO로서 존경 또한 한 몸에 받고
계신 듯 보여집니다. 많이 겪어 보지는 못했지만 말씀 하나 하나 하실 때 단어선택에도 신경을 쓰셔서 말씀을 하시고 한마디
한마디에 진심을 담아 나아갈 비젼을 저희에게 보이시고 우리의 생각을 물으시고 들어주심에 회사의 직원분들이 왜 이토록 친절하시고
배려가 많으신지 알 수가 있었습니다. 저는 이것을 끝으로 여기지 않습니다. 지금은 다시 학교로 돌아가지만 분명 회사에서 만족할
만한 인재가 되어 다시 이곳을 방문했을 때는 이렇게 아쉬운 마음으로 돌아가지 않을 것입니다. 그때가 너무 기다려지고 기대가
됩니다. 다시 한번 귀한시간 내어 저를 4주가량 맡아서 친절과 배려로 가르쳐주신 인실리코젠의 모든 직원분들에게 감사의 말씀을 짧게나마 드리면서 인실리코젠과 함께한 4주간 “人Co” 인턴십에 대한 후기를 맺겠습니다. 감사합니다!
김진규
[첫 만남 : 2013년 1월 6일 (일)]
人Co
Internship의 첫 시작은 1월 7일 월요일이었지만, 내게 있어서 실제 인턴십의 시작은 1월 6일 일요일이었다. 수원
사무실에 8시까지 출근해야 된다는 사실을 알게 되었고, 평소 잠이 많은 나는 고민 끝에 4주 동안 수원 사무실 근처에서 살기로
마음 먹었다. 한 달간 일하게 될 인실리코젠 본사에도 미리 와보고 주변 고시원, 숙소들도 알아보았지만 마땅치 않았다. 게다가
6개월도 아닌 한 달 동안 살 방을 알아보는 것은 더욱이 어려웠고 수원역 주변은 이른 아침마다 출근하기엔 교통편에서 별 이점이
없어 보인다고 생각하여 지나가던 중 찜질방을 보게 되었고, 나는 1월 6일부터 2월 1일 한 달간 찜질방에서 살게 되었다. 사실
처음부터 찜질방 생활이 편했다고 할 순 없지만 약 4주가 된 지금은 정말 편하게 잘 지내고 있다. 다음 날 나는 인실리코젠에
처음으로 출근을 하게 되었다. 지문 등록, 위키 문법과 OJT작성, 사내 예절 교육, Skype 등 모든 것이 처음 접해보는
것이고 낯설었고 어려웠다. 다른 인턴 분들께서 먼저 와 계셨는데 당시는 정말 어색하고 긴장됬지만 한 주 한 주 같이 배우고 일하고
발표하면서 정말 큰 힘이 되었다. 만약에 다른 인턴분들이 없었다면 지난 4주 동안 어떡했을까 하는 생각이 든다.
[1주차]
첫
날 이후 인실리코젠 직원 분들을 익히는 것이 어려웠다. 그 이유는 직책이 너무 다양했기 때문이다! 이전에도 아르바이트 등을
해왔고, 군대에서도 금방 익혔는데… 선임님, 주임님, 책임님, 수석님….너무 헷갈렸던 기억이 난다. 뿐만 아니라 그 동안 가끔씩 한
두 번 만들어봤던 프레젠테이션 작성과 발표를 매주 2번 정도씩 발표 혹은 제출해야 된다는 사실을 알게 되었다. 그야말로 1주차
초반에는 정말 매일 매일 혼란의 연속이었다. 첫 발표는 수요세미나 시간에 했었던 “자기소개” 였다. 첫 발표라 많이 긴장도 많이
됐지만 그것보다도 앞으로의 발표를 위해서 정말 많이 연습하고 준비해야겠다는 생각이 앞섰다. 이후 성격 검사, 사내 예절,
농림수산검역검사본부 세미나 보조 등 그렇게 첫 1주는 정신 없이 지나갔다. 사실상 4주라는 기간 동안 생물정보 분석 프로그램을
완전히 익히는 것은 어렵기 때문에 2주차에는 당뇨에 대한 Case study를 실습해보기로 하였고 3주차에는 인플루엔자 바이러스에
대한 Case study를 실습하기로 하였다. 그리고 마지막 4주차에는 NGS 분석기능이 추가된 CLC Genomics
workbench에 대해서 공부를 하기로 하였다.
[2주차]
2주차에는 본격적으로 인실리코젠의 생물정보 분석 메인 툴 중 하나인 CLC Main Workbench에 대해서 공부를 하였다.
처음인지라 CLC Main workbench를 다루는 것 자체가 서툴고 어떠한 기능을 언제 활용 해야 될 지도 감이 안 왔지만,
김경윤 주임님과 송하나 선배님을 비롯, 다른 인턴분들에게 조언을 구하면서 Case study를 실습한 끝에 어느정도 전반적인
내용은 이해할 수 있었다. 처음에는 어려웠지만 Case study를 통해 실습을 해보면서 한 가지 실험을 다루면서, Main
workbench에 대한 기능도 통합적으로 공부할 수 있어서 정말 효과적이었다고 생각한다. 학교 전공과목 시간에 이론적으로 배웠던
SNP, BLAST 등의 내용을 실제로 프로그램을 이용하여 실습하고 관련 내용을 찾아서 공부해나가다 보니 정말 내 자신에게 큰
도움이 되었다. 실습도 점점 익숙해 졌을 뿐 아니라 첫 주와는 달리 회사 분위기도 빠르게 적응이 되었다. 한 분 한 분 모두들
정말 친절하시고 열정을 가지고 일하시는 모습이 인상 깊었다. 비록 금요일에 발표는 만족스럽지 못했지만 어느 정도 Main
workbench에 대해서도, 인실리코젠에 대해서도 감을 익힐 수 있었던 한 주였다.
[3주차]
어느덧 人Co Internship 프로그램에 참여한 지도 3주차, 벌써 절반이 지났다. 이번 주에 다루게 될 CLC Main
Workbench의 2번째 Case study는 인플루엔자 바이러스에 대한 연관성을 분석하는 내용이었다. 몇 해전 전세계적으로 큰
이슈였던 조류 독감, H5N1
인플루엔자 바이러스들의 지역별, 종별 연관성을 비교하는 실습을 하였다. 뿐만 아니라 유사 질병인 뉴캐슬 병과의 차이점도 CLC
Main Workbench를 이용하여 손쉽게 비교할 수 있다는 사실에 정말 신기했다. 처음 CLC Main Workbench를
이용할 때는 마냥 어려운 프로그램이라고만 생각했었는데 어느 정도 익숙해지니 정말 생물정보 분석을 하는데 유용한 툴이라는 생각이
들었다. 중간 중간에 틈틈이 최남우 대표이사님, 임천안 이사님, 박준형 부장님과 면담을 가졌는데 내 자신의 현재를 다시 한번
되돌아 보고 반성할 수 있었던 좋은 기회였다. 뿐만 아니라 아직은 불확실한 진로에 대해서도 좀 더 많이 고민하고 방향을
잡아나가야겠다는 생각이 들었다. 금요일 발표와 함께 그렇게 인턴십의 3주차도 지나갔다.
[4주차]
벌써 인턴십의 마지막 주가 되었다. 지난주부터 틈틈이 공부해온 NGS에 대한 배경지식을 CLC Genomics workbench를
이용하여 인턴분들 각각에게 주어진 다른 주제를 다뤄보았다. 나는 Epigenomics와 ChIP-chip/ChIP Seq 두 가지
분석 방식을 알아보고 실제로 예제 데이터를 이용하여 ChIP Seq 분석을 해보았다. 기존의 Main workbench에 NGS
데이터 분석 기능이 추가되었기 때문에 이해하는데도 좀 더 어려웠지만 많은 분들의 도움을 받아 무사히 프로젝트를 수행하고 발표를
마쳤다.
[2013년 2월 1일 (금)]
오늘은 드디어 人Co Internship의 마지막 날이다. 오전에는 그동안 4주간의 인턴십 활동을 집약하여 만든 프레젠테이션을
토대로 최종평가 발표 시간을 가졌다. 최남우 대표이사님과 임천안 이사님, 정은미 이사님, 김경윤 주임님 그리고 이지혜 주임님께서
평가관으로 참석하셨는데 너무 떨리고 긴장됐다. 나머지 세 분의 인턴분들의 발표까지 다 끝난 이후 대표이사님께서 여러 가지 조언과
함께 인실리코젠의 경영 철학인 人Co에 대해서 다시 한번 말씀하셨는데 다시 한번 이번 인턴십 기간 동안 배우 많은 것들의 의미를
되새길 수 있었다. 오후에는 수료식을 가졌는데 4주라는 기간, 총 160시간 동안의 人Co Internship 동안 직접 경험한
모든 순간 순간들이 새록 새록 기억나는 순간이었다. 인턴십 기간 동안 매일 작성해온 OJT 위키 페이지에 랜덤으로 나오는 글귀 중
이런 글귀가 있다.
“엘리자가 말했어요. 세상은 생각대로 되지 않는다고. 하지만 생각대로 되지 않는다는 건 정말 멋지네요. 생각지도 못한 일이 일어나니까요."
그렇다. 항상 무언가를 준비하고 직접 실천하는 데 있어서 나 자신의 생각대로 완벽하게 되는 것은 없다. 하지만 그렇기 때문에 보다
많은 것을 경험하고 배울 수 있으며 다시 한번 자신에 대해서 되돌아 볼 수 있는 여유를 가질 수 있는 것이 아닐까? 나는 이번
人Co Internship을 통해서 다시 한번 내 자신의 부족한 점이 무엇이고 앞으로 보완해 나가야겠다는 생각이 들었는데
이것만으로도 나는 인실리코젠에서 귀중한 경험을 했다고 생각하고 앞으로도 잊지 못할 것이다.

유승일
Insilicogenist로 지낸 1월
(주)인실리코젠에 대한 첫 기억은 2010년 말 송하나 선배님이 취직했다는 소식을 접했을 때 였습니다. 그 이후엔 졸업생
선배들의 입에서 저희 회사에 대한 얘기를 은연 중 들었던 기억도 있습니다. 그렇게 한두 번 회사명을 접하다보니 '회사명 정말
좋다.' 라는 생각을 하게 되었습니다. in silico + gen(e), 누가 들어도 생물정보회사였습니다.
석사 졸업을 앞 둔 시점에서 첫 직장에 대한 고민을 하게 되었습니다. 항상 '첫 직장은 중요하다.'라는 말을 들어왔기 때문인지 그
어느 때보다 신중했던 것 같습니다. 선택을 하게 된 큰 이유는 주변 지인들의 자자한 칭찬이 있었기 때문입니다. 사내 분위기도
좋고, Culture Day를 통해 단합을 다지는, 사람이 중심인 회사라는 칭찬이 그것이었습니다. 하지만 제 선택의 주요인은 회사
홈페이지를 봤을 때 부터 마음이 이미 넘어와 있었습니다. 그리곤 人Co Internship의 문을 두드렸습니다.
7일부터 8일 이틀간은 인코인턴십 프로그램의 소개와 저희 회사에서 개발한 LabKM을 기반으로 한
Wiki의 사용법, 그리고 人Co 브랜드 가치 소개, 사내 생활에서의 예절, 안전보안교육 등 회사생활에 필요한 다양하고도
기초적인 교육을 받은 시간이었습니다. 이 시간 동안 명함을 주고받아 보기도하고 제 성격이 어떠한 유형인지 알아 본 시간이
되었습니다. 많은 교육들 중에 제 머리 속에 딱 박히는 단어가 있었습니다. 바로 정은미 이사님께서 말씀해주신 Plan Do See
의 생활화 입니다. 이사님께서는 회사를 다니시면서 박사학위과정을 밟으셨는데, 그때 일을 말씀해주시면서 Plan Do See 라는
말씀을 해주셨습니다. 이 방법은 제가 그 동안 해왔었던 것들을 다시 한 번 돌아보는 계기가 되었습니다. 또한 이번 1월 한 달
동안 프로그램을 진행하면서 직접적인 큰 도움이 되었습니다.

그 다음 날인 9일에는 가장 떨렸었던 첫 발표인 자기소개가 있었던 날입니다. 수요세미나 때
진행되었기 때문에 스카이프를 통해 모든 직원분들에게 저에 대한 간략한 소개를 하는 시간이었습니다. 석사학위논문에 대해서도
말씀드리기도 했지만, 저의 포부를 보여드리기 위해 ‘하나로 잇는 유승일’, ‘유승일-INSILICOGEN=0'이라는 멘트를
준비했었습니다. 발표 당시에는 당차게 발표했었는데 지금 생각하면 얼마나 손발이 오글거리는지 모릅니다.
본격적인 일정이 시작되었던 10일에서 23일에는 CLC Main Workbench에 대해서
공부했습니다. 당뇨와 조류 인플루엔자, 총 2가지 케이스에 대해 매뉴얼을 참고하며 Main Workbench를 익히는 시간이
되었습니다. 당뇨병 케이스 스터디에서는 SNP가 당뇨병에 어떻게 연관을 보이는지에 대해서 공부했습니다. SNP에 의해 단백질의
구조가 바뀌면 그 기능도 변하기 때문에 Main Workbench를 통해 당뇨병을 유전적인 레벨에서 공부하는 계기가 되었습니다.
두번째 케이스로 조류 인플루엔자에 대해서 공부했었습니다. 조류 인플루엔자는 바이러스가 원인인데 고 병원성 바이러스에 걸리면 엄청난
전염성으로 인해 막대한 경제적 손실을 가져옵니다. 때문에 공부를 하면서 고 병원성과 저 병원성의 염기서열의 차이를 밝히면
재밌겠다는 아이디어가 떠올랐지만, 논문 검색을 해본 결과 아쉽게도 다른 사람이 먼저 연구한 것이 확인되었습니다. 어쩔 수 없이 그
논문의 내용을 가지고 cleavage site에서 고 병원성과 저 병원성의 서열 차이를 Main Workbench를 사용하여
확인했습니다.
24일부터 30일에는 Main Workbench를 마치고 Genomics Workbench를
공부하였습니다. NGS 데이터를 활용한 Genomics Workbench의 다양한 활용 중에 저는 Expression
Analysis를 공부했고 분석을위해 RNA-Seq을 Genomics Workbench를 통해 수행했습니다. 분석을 공부하면서
느꼈던 것은 효율적인 시퀀싱 컨설팅 진행을 위해서는 실험 방법이나 과정을 제대로 이해하는 것부터 시작되어야 하고, 유전학적 지식을
바탕으로 대용량 시퀀싱 데이터를 효율적으로 활용할 수 있는 기반이 체계적으로 구축되어야 한다고 생각했습니다. 프로그램의 자동성과
스크립트를 통한 유연성을 골고루 갖추는 것이 좋을 것 같았습니다.
중간 중간 독서경영과 추가적인 업무들을 포함해 Genomics Workbench를 마지막으로 1월 한 달의 모든 과정이
끝났습니다. 그 한 달 동안 저는 39번의 지하철과 78번의 버스, 그리고 78번의 환승, 2666.43km의 이동 그리고
97시간 30분의 출퇴근 이라는 기록 데이터를 만들어냈습니다. 이 기록은 계속 이어질 테지만 단순히 이어지는 것이 아닌 이 기록을
밟고 더 나아가야 할 것은 프로페셔널단계입니다. 프로가 되고 싶다. 이것이 제가 한달 동안 人Co Internship을 하면서 가장 크게 느낀 점입니다.
한 달 동안 진행된 人Co Internship은 제 인생에 특별한 의미가 있는 달이었습니다. 제 인생에 있어서 화이트
칼라(White Collar)로써 처음으로 출근을 경험한 의미 있는 달이고, 진정한 아침형 인간으로 환골탈태한 달이기도 합니다.
그리고 저만의 아날로그 식 메모가 유원기 주임님 덕분에 스마트한 메모로 탈바꿈하기도 했습니다. 그리고 부족한 부분이 무엇이고,
어떻게 채워 나가야하는지 나름의 방향을 잡는 계기가 되었고, 인실리코젠이 컨설팅전문회사로써 어떤 업무를 하는지 100%는 아니지만
어느 정도 알게되었습니다. 1월의 人Co Internship은 끝이 났습니다. 하지만 앞으로 2월의 人Co Internship
그리고 앞으로 차근차근 계획하고 실천하고 평가하면서 제 역량들을 쌓아갈 것입니다. 마지막으로 좋은 기회를 주신 사장님과 직접
실천으로 가르침을 주신 모든 분들께 감사드립니다.

Posted by 人Co









 입사지원서_2014.docx
입사지원서_2014.docx
 입사지원서.docx
입사지원서.docx
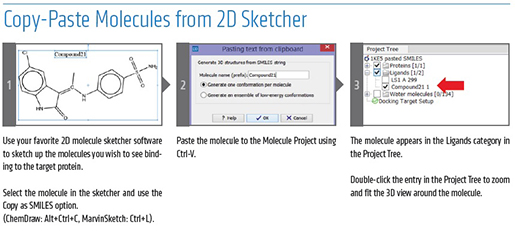
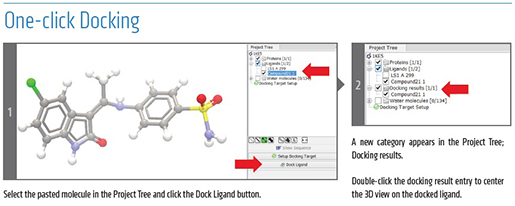
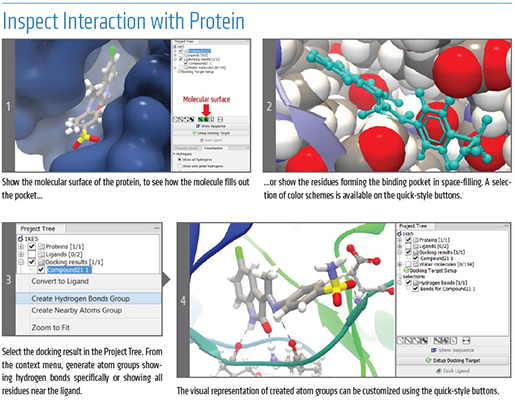

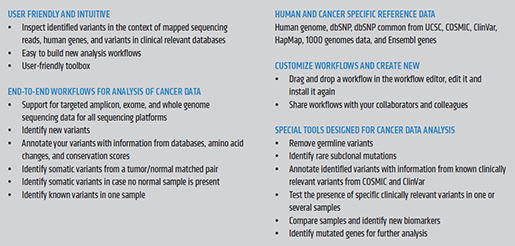
 CLC_DDW_ProductSheet.pdf
CLC_DDW_ProductSheet.pdf