<온라인몰 오픈기념> CLC Main Workbench 프로모션 당첨자 발표!
- Posted at 2015/10/20 15:00
- Filed under 회사소식
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/192















Posted by 人Co
(The power of the Web is in its universality, Access by everyone regardless of disability is an essential aspect.)
팀 버너스 리 경 - 웹의 창시자 (Tim Berners - Lee , W3C Director and inventor of the World Wide Web)
웹 접근성은 2008년 '장애인차별금지 및 권리구제 등에 관한 법률(이하 장애인차별금지법)'이 제정된 이후로 국내에서도 그 법률적 근거가 마련되었고, 2013년에 이르러 민간 기업까지 그 적용 범위가 확대되었습니다. 한국정보화진흥원 웹 접근성 연구소에서 제시하는 웹 접근성 정의는 다음과 같습니다.
위 정의에 따르면 웹 접근성은 특정 대상에 한정되지 않고 모든 사용자를 대상으로 하며 모두를 유익하게 하는데 그 목적이 있습니다.
웹 접근성에 대한 관심과 필요성에 대한 인식이 커지고 있지만 그 정의와 범위가 모호하고 방대해 실무자에게는 아직까지 와 닿지 않거나 어렵게 느껴지는 것이 현실입니다. 이 에 웹 콘텐츠 접근성 지침을 위한 국가 표준으로 KWCAG(Korea Web Content Accessibility Guidelines, 한국형 웹 콘텐츠 접근성 지침) 2.1이 마련되었고 장애인이 비장애인과 동등하게 웹 콘텐츠에 접근할 수 있도록 웹 콘텐츠를 제작하는 지침 및 방법에 관하여 기술하고 있습니다. KWCAG 2.1은 원칙(Principle), 지침(Guideline), 검사항목(Requirement)의 3 단계로 구성되어있고 각 각의 내용은 웹 접근성 제고를 위한 4가지 원칙과 각 원칙을 준수하기 위한 13개 지침 및 해당 지침의 준수 여부를 확인하기 위해 22개의 검사항목으로 구성되어 있습니다.
웹 접근성에 대한 실무적 접근을 돕고자 웹 콘텐츠의 접근성 준수여부를 평가해 볼 수 있는 도구들을 소개하려 합니다.
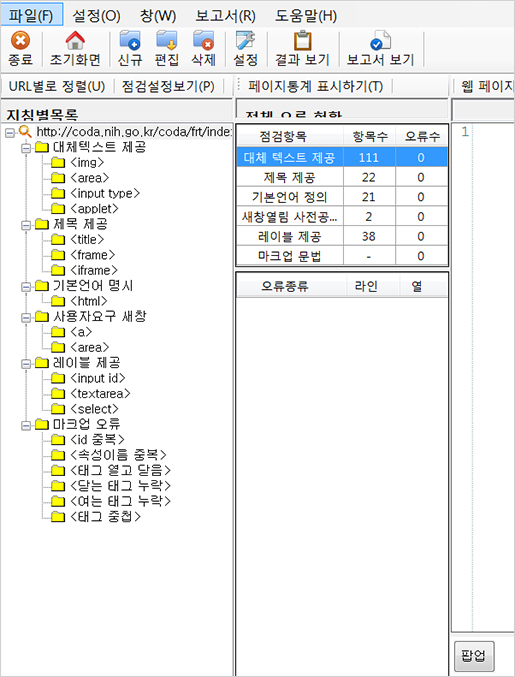
다운로드 및 설치
1. 웹접근성 연구소 접속
2. K-WAH 4.0 프로그램 내려받기 
3. 파일을 다운받고 원하는 위치에 압축을 푼 후 파일 실행
4. 사용권계약에 동의하고 경로를 설정한 후 설치
5. 설치를 완료하면 바탕화면에 'K-WAH 4.0 바로가기' 아이콘 생성
진단가능한 검사항목
OpenWAX
Firefox 부가기능으로 제공되고 있는 소프트웨어로 개별 페이지별로 웹 접근성 준수여부를 확인할 수 있습니다.
* 위 이미지는 2015년 당사에서 개발 및 웹접근성 인증마크를 획득한 바 있는 의과학지식센터 웹사이트를 테스트하여 캡쳐한 이미지입니다.
다운로드 및 설치
1. 파이어폭스 부가기능 으로 이동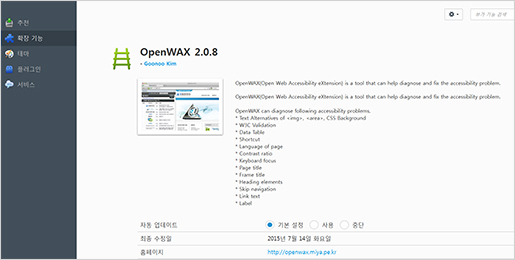
2. '+Firefox에 추가' 버튼 클릭
3. 소프트웨어 설치 창에 [설치] 버튼 클릭
4. Firefox 다시 시작
5. Firefox 주소창 우측에 사다리모양 아이콘 생성
사용방법
1. OpenWAX 아이콘(사다리모양) 클릭
2. 사이드바에 OpenWAX가 실행되면 [현재 페이지 검사] 버튼 클릭
3. 검사항목별 검사결과 확인
진단가능한 검사항목
Colour Contrast Analyser
전경색과 배경색의 명도 대비를 확인할 수 있습니다.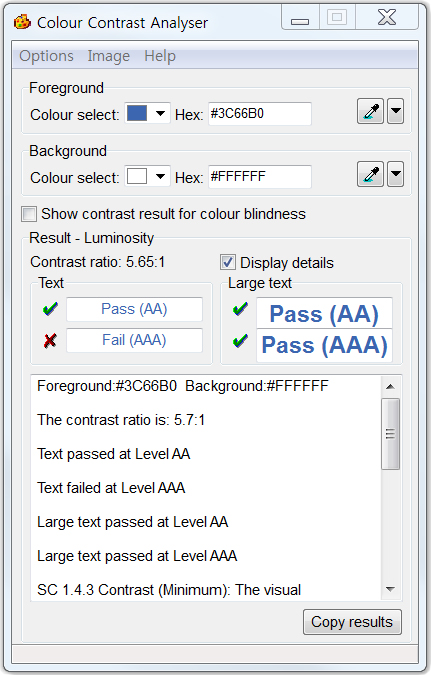
다운로드 및 설치
1. 다운로드
2. 다운로드 받은 압축 파일을 압축 해제하여 압축 해제된 폴더를 적당한 위치로 이동
3. 폴더 내의 Colour_Contrast_Analyser.exe 파일을 실행
작성자 : 데이터사이언스센터 프론트앤드개발자
김근래 주임
Posted by 人Co

• 일시 : 2015년 10월 12일(월)~ 10월 14일(수)
• 장소 : KT인재개발원 1연수관 207호
미생물 유전체 분석에 대한 전반적인 이해와 분석 전략 및 실습
(자세한 프로그램 내용은 http://kobicedu.labkm.net 참고)
신청방법
• 신청기간 : 2015년 9월 16일(수) ~ 2015년 9월 25일(금)
• 선발인원 : 30명
• 교육대상 :
1) 미생물 유전체 분석에 대한 이해와 분석 방법의 교육이 필요한 연구원 및
대학원생 등
2) 모든 교육 일정에 참석이 가능한 교육생 (3일 일정 필수 참석)
• 선발안내 : 2015년 10월 1일(목) ~ 2015년 10월 2일(금)
• 교육비 : 무료 (중식 무료제공)
• 준비물 : 유무선 인터넷이 가능한 개인 노트북
• 신청방법
- 온라인 신청 http://kobicedu.labkm.net
• 문의
- ㈜인실리코젠 (![]() 031-278-0061, edu@insilicogen.com)
031-278-0061, edu@insilicogen.com)
- 문의게시판 이용 http://kobicedu.labkm.net/labboard/board/QnA
Posted by 人Co

ISCB(International Society for Computational Biology)에서 주최하는 ISMB(The 23rd Annual International Conference on Intelligent Systems for Molecular Biology)가 2015년 7월 11일부터 14일까지 4박 5일 동안 아일랜드 더블린의 Dublin Convention Centre에서 개최됐다. 이번 ISMB는 ECCB(The 14th Annual European Conference on Computational Biology)와 함께 열려 더욱 풍성한 내용을 담은 교류의 장이었다. ISMB와 ECCB는 bioinformatics와 computational biology, genomics, computational structural biology는 물론 system biology를 포함한 공통의 관심사를 갖는 국제학회이기 때문에 2004년부터 매 2년 마다 학회를 함께하고 있다. 2017년에 열릴 ISMB/ECCB는 체코의 수도인 프라하에서 열린다고 하니 국내외 연구자들과 세계적으로 아름다운 도시에서 지식을 나눈다는 것이 기대된다. 이번 ISMB/ECCB2015가 열린 아일랜드의 더블린 또한 역사와 전통이 깊은 도시로 많은 기대를 품고 참석했다.
ISMB
ISMB convenes an interdisciplinary group of scientists dedicated to the advancement of biological discovery through computation
ISMB educates scholars at all stages of their career
ISMB showcases state-of-the-art advances in the dynamic fields of computational biology and bioinformatics
ISMB is the forum for introducing new directions and for announcing technological breakthroughs
ISCB

5일 동안 진행되는 이번 ISMB/ECCB2015의 일정은 크게 pre-conference(July 10-11, 2015)와 main-conference(July 12-14, 2015) 두 가지로 나뉜다. Pre-conference 기간 동안에는 SIGs(Special Interest Groups) 미팅이 진행되며 이 미팅은 총 9개의 세부 모임으로 구성된다(Table 1). 각각의 모임은 등록기간 내에 신청하고 일정량의 금액을 미리 지불해야한다. 이번 학회의 SIGs 모임은 BioVis와 HiTSeq을 신청해 참가했는데 BioVis에서는 주로 pathway, GO, comparative genomics에 대한 visualization을 다뤘고, HiTSeq 에서는 NGS를 통한 다양한 응용연구분야에 대한 톡이 주를 이뤘다.
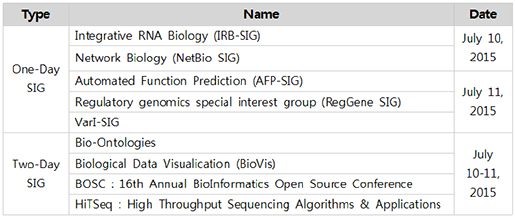
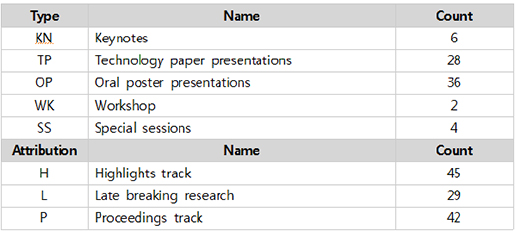

또한, 각 세션마다 테마를 설정해 학회 참석자들에게 선택의 편의성을 제공한 점은 작년(2014년)과 다른점이다. 테마는 GENES, DISEASE, PROTEIN, SYSTEMS, DATA, OTHERS로 구성되어 있다.
July 12-13 저녁시간에는 저자와 학회 참석자가 함께 자유로운 토론을 할 수 있는 포스터리셉션이 진행됐다. (주)인실리코젠도 non-model species analysis를 주제로 두 편의 포스터와 함께 참가했다. ISMB/ECCB 학회의 특성상 system biology 분야가 주를 이루기 때문에 non-model species analysis에 대한 큰 관심을 기대하지 못했다. 하지만 많은 국외연구자들이 non-model species에 대한 관심을 갖고 있었고 non-well model species의 re-annotation에도 큰 관심을 갖고 있었다. 질의 중 새로운 관점을 느꼈는데 transcriptome analysis에서 de novo assembly와 expression abundance를 계산할 때 샘플링 단계에서 total mRNA를 취할 것이냐, single cell을 취할 것이냐에 대한 것이었다. 최근 분석 트렌드는 single cell에서의 development 등을 확인하는 것인데 그것을 염두한 의견같다. Single cell이 pooling cell을 커버할 수 있을 것인가와 pooling cell에서 missing point가 생기지 않을까에 대해 나눴고, 보고자 하는 연구 목적에 따라 다르다는 결론을 내렸다.
참가 포스터 1제목 : An integrated pipeline and monitoring system for de novo genome analysis [F09]
저자 : Junhyung Park, SeungJae Noh, Kyuyeol Lee, Yeonkyung Kang, Myunghee Jung

제목 : De novo transcriptome assembly and in silico expression PROFILES of Sebastes schlegeli [E41]
 Figure 2. Exhibitors of ISMB/ECCB2015
Figure 2. Exhibitors of ISMB/ECCB2015
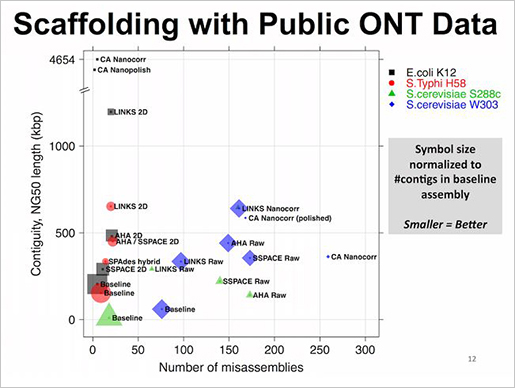
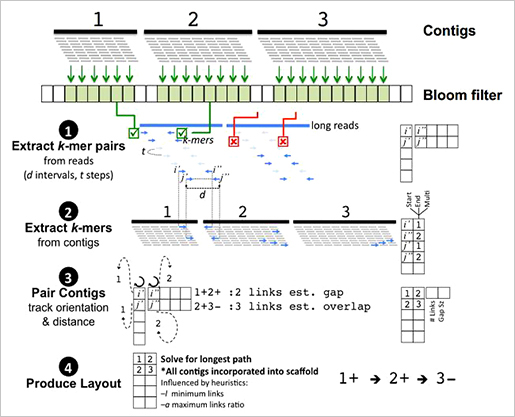
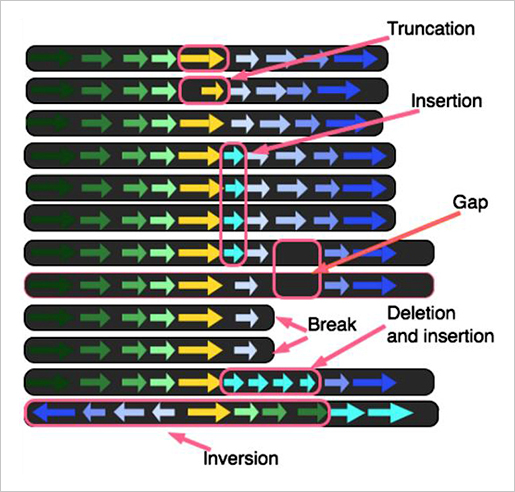
LINKS는 메모리 효율성이 매우 뛰어나다. 그 이유는 scaffolding algorithm에 있다. long reads를 짧은 k-mer pairs로 추출하고 scaffolding의 대상이 되는 contigs도 k-mer pairs를 추출한다. 각각에서 추출된 k-mer pairs의 서열상동성이 같은 위치정보와 paired-end information을 통해 scaffolding을 수행한다. 또한 iteration 수를 높게 조절함으로써 정확도를 향상할 수 있다는 장점도 존재한다.


UniProt

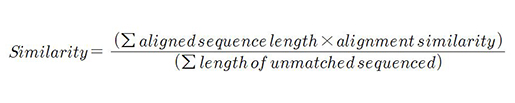
매우 많은 entry가 줄어서 데이터베이스의 크기 자체는 줄었지만 훨씬 specific하고 meaningful한 데이터베이스가 탄생했다. 줄어든 데이터베이스는 ordering을 통해 여러 개의 component로 merge되어있다. 현재 UniProt site에 released database는 proteome redundancy removal 버전이다.

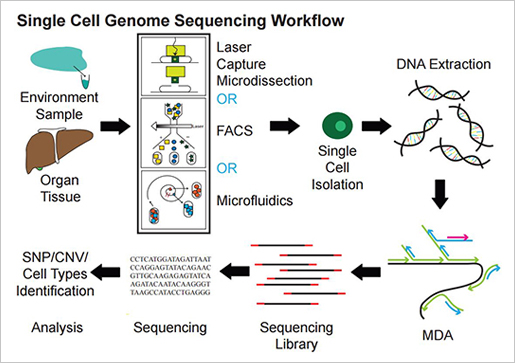
국내 연구동향 및 시퀀싱 회사의 single cell analysis는 아직 걸음마 단계에 불과하다. Single cell에서 DNA와 RNA를 분리해 시퀀싱하고 그 안에서 일어나는 생물학적 이벤트는 epigenomics 연구는 물론, cell cycle에 따른 cell-to-cell correlations, T cell 연구 등 다양한 분석에 적용이 가능하다.
Genotyping By Sequencing (GBS)는 차세대 시퀀싱 기술을 바탕으로 새롭게 개발, 발전하고 있는 NGS 분석법 중에 하나이다. 유전체 전체를 시퀀싱하는 WGS에 비해 저렴한 비용으로 빠르고 쉽게 genome-wide 분석을 가능하게 한 테크닉이다. 제한효소를 처리하여 유전체 서열에서 그 제한효소에 의해 잘리는 영역 주변의 서열만을 시퀀싱하게 된다. 제한효소를 처리한다는 점에서는 RAD-seq과 근본적으로 원리가 같지만 효소절단 후 사이즈 선별을 하지 않는다는 점에서 시퀀싱 라이브러리 제작이 보다 간단한 편이며 GBS 시퀀싱 데이터가 RAD-seq에 비해 low coverage로 얻어진다.
GBS는 아래와 같은 applications이 있다.

작년 ISMB2014에서는 de novo assembly 나 expression analysis, GO, KEGG, COG, pathway 연구와 같은 전반적이고 일반적인 연구가 상당수를 차지했다. 하지만 올해의 ISMB학회는 ECCB와 함께했기 때문인지 그런 비율이 많이 줄었고 좀 더 세분되고 집중적인 연구가 주를 이뤘다. 특히 암과 같은 특정 질병의 원인 판별과 치료법 개발에 초점이 맞춰져 있었다. 세션들의 주제가 GENES, PROTEINS, SYSTEMS, DATA 이외에 DESEASE도 있다는 것은 이를 반증하고 있으며, DESEASE 세션의 개수도 상당하고 그 연구수준도 점진적으로 발전되고 있어 보인다. 특히 연구방법들이 한가지 방향으로 모이지 않고 다양한 시도를 통한 기초연구에 힘쓰는 것에 놀랐다. 이는 한국의 연구풍토와 유럽, 미국의 연구풍토가 많이 다르기 때문으로 생각한다. 언제쯤인지 퇴근길에 읽은 어느 연구자님의 문구에 따르면, 정확한 메커니즘의 이해보다는 응용성을 강조하는 우리나라의 스타일과는 다르게 유럽이나 미국은 정확한 원리를 이해하고 그것을 수학적인 도구를 통해 분석해야 하며 실험의 결과들이 높은 재현성을 나타냄과 더불어 각각의 결과들이 강한 유기성을 가질 때 비로소 올바른 결과로써 인정한다고 한다. "블루오션은 찾아내는 것이 아닌 만드는 것이다.", "소비자는 자신이 무엇을 원하는지 모른다."는 명언들이 말하는 창조적 사고의 연구풍토가 선진국이 될 한국에도 자리 잡을 때가 아닌가 생각한다.
NGS의 도입과 함께 전체를 아우르는 분석이 가능해졌고 새로운 그 무언가를 찾을 기회가 많아졌다. 작년까지는 이런 NGS의 장점과 특성을 살린 연구추세였다면 점차 NGS의 응용범위가 좁은 범위까지 확대되어가는 중이다. Iontorront에 이어 nanopore와 같은 소형 시퀀싱머신의 개발과 보급이 일반화되기 시작했다. 앞으로는 국내의 실험실에도 많은 변화가 있을 것이며, 그 중심은 또 한 번 NGS가 될 것이다. 이젠 국내에서도 bioinformatics의 인식이 달라져야 한다.
Posted by 人Co
