SARS-CoV-2 바이러스 단백질을 선택된 네트워크에 연결하는 탐색 도구, Coronavirus Network Explorer
- Posted at 2020/07/17 12:48
- Filed under 지식관리

전 세계 COVID-19 확진자가 1,000만 명을 넘어선 지금, 일일 확진자 수의 상승세와 함께 한국에서도 COVID-19는 재유행 국면을 맞고 있습니다. FDA에서는 아직 백신과 치료제가 없다고 공표하였고, 의료진의 처방 이외의 약품에 주의하라고 권고하고 있습니다.
SARS-CoV-2를 잘 이해하기 위해, 이들이 인간의 숙주 단백질에 어떤 영향을 미치는지 예측된 생물학적 네트워크를 확인할 수 있는 웹사이트인 Coronavirus Network Explorer를 소개합니다.

Coronavirus Network Explorer는 SARS-CoV-2가 숙주 세포에 어떤 영향을 미치는지 보여주는 생물학적 네트워크를 살펴볼 수 있는 웹페이지입니다(Fig.1). 생물/의학 문헌의 증거를 바탕으로, 이 네트워크들은 대규모 지식 그래프 및 머신러닝을 사용하여 선택된 생물학적 과정, 질병 또는 경로에 대한 유전자 및 약물 영향을 예측합니다. 지금부터 Coronavirus Network Explorer를 소개하려고 하며, Fig.1에서의 위치(좌측/우측 등)를 기준으로 설명하고자 합니다.

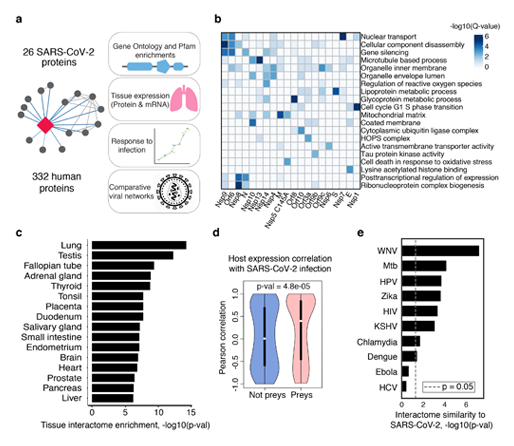
최근 Gorden 외 연구진은 친화 정제-질량 분석법(AP-MS)을 통한 스크리닝으로 SARS-CoV-2 바이러스 단백질과 상호작용하는 인간 숙주 단백질을 확인하였습니다(출처 : Gordon et al., 2020. Nature.). COVID-19에 대비하여 잠재적으로 사용할 수 있는 약물을 확인하기 위해 숙주 단백질은 기능적으로 특징지어지고, 기존 약물 표적에 대해 스크리닝 되었습니다(Fig.2). 생물/의학 문헌에서 얻은 지식 데이터베이스인 QIAGEN Knowledge Base와 머신러닝이 결합한 IPA를 바탕으로(출처 : Krämer et al., 2013. Bioinformatics.), Gorden 외 연구진의 연구결과인 SARS-CoV-2와 숙주 단백질과의 상호 작용 정보를 바이러스 감염에 의해 영향을 받는 생물학적 기능 또는 질병에 연결되어 네트워크가 구성되었습니다.

Coronavirus Network Explorer에서 분석할 수 있는 것은 크게 다음과 같습니다.
- SARS-CoV-2가 숙주에 미치는 영향을 설명하는 네트워크 탐색
- 설정한 조건에서 유전자의 예측된 활동 패턴 표시
- 바이러스의 효력에 영향을 줄 것으로 예상하는 약물의 잠재적 표적 유전자를 식별
Coronavirus Network Explorer의 구성을 자세히 살펴보도록 하겠습니다.
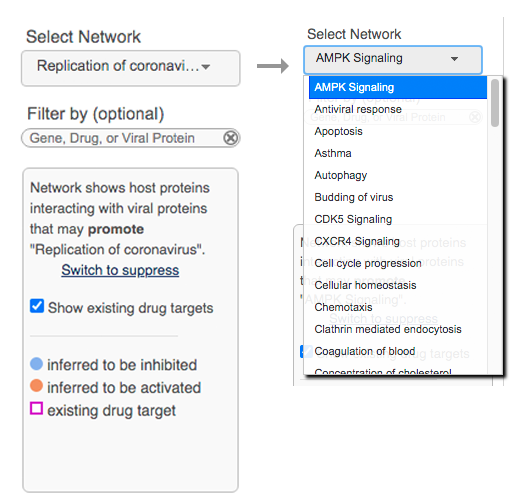
[Fig.3] (좌) 좌측 패널 구성, (우) Select Network 패널의 구성
Coronavirus Network Explorer는 좌측 패널의 조정을 통해 원하는 네트워크를 도출하게 됩니다. 사용자가 원하는 설정을 할 수 있는 부분(빨간색)과 네트워크의 촉진/억제에 관련된 설명(파란색), 그리고 각 노드의 색에 대한 설명이 있는 부분(초록색)으로 나뉘게 됩니다(Fig.3).
Select Network 패널에서는 SARS-CoV-2와 인간의 단백질이 연관되어 있다고 예측된 네트워크들을 확인할 수 있습니다(Fig.4).
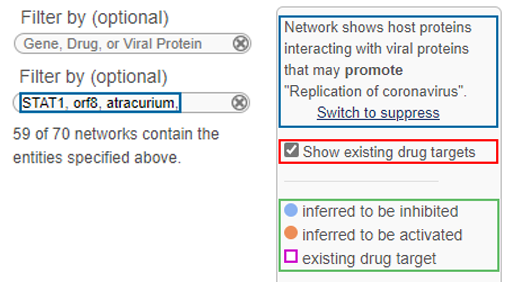
[Fig.4] (좌) Filter by 패널의 구성, (우) 네트워크에서 보이는 legend 정보
앞서 말씀드린 두 패널 아래에는 숙주 단백질과 상호 작용하는 바이러스 단백질에 의해 현재 보고 있는 해당 network가 촉진/억제되는 것으로 추측되는 결과라는 것을 알려줍니다(Fig.6, 파란색). 또한, 기존 약물 표적 유전자를 표시할 것인지의 여부와(빨간색) 억제/활성으로 추론되는 네트워크상 구성 노드의 표시 및 존재하는 약물 표적을 확인할 수 있습니다(초록색).
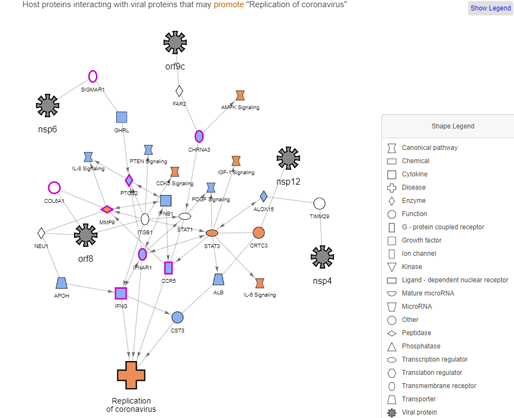
화면 가운데 영역은 각 패널에서 설정한 네트워크를 보여줍니다(Fig.7). Fig.7은 예시로, "Replication of coronavirus"라는 네트워크 정보입니다. 노드마다 생김새가 다른 것을 볼 수 있는데, Show Legend 버튼을 클릭하면 이 노드들이 어떤 기능을 하는지 확인할 수 있습니다. 노드들은 클릭 후 드래그를 통해 이동시킬 수 있습니다.
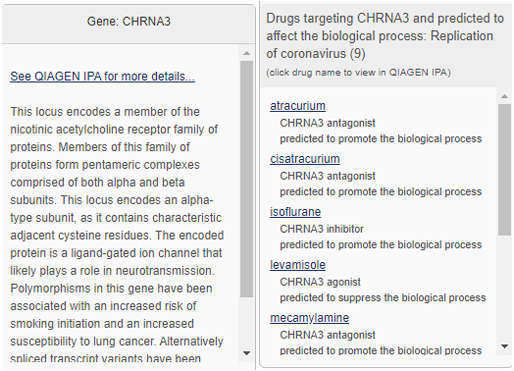
노드를 클릭하면 해당 노드에 관련된 정보를 화면 우측 영역에서 확인할 수 있습니다(Fig.8). 해당 영역에서 유전자 정보(왼쪽), 기존 약물 표적 단백질의 경우 약물 리스트를 볼 수 있습니다(오른쪽). Fig.8은 Replication of coronavirus 네트워크상의 CHRNA3 노드를 클릭한 결과입니다. IPA 사용자들은 더 자세한 유전자 정보와 약물 정보를 링크 클릭으로 확인할 수 있습니다.
이처럼 SARS-CoV-2의 단백질과 인간의 단백질이 상호작용하는 정보를 바탕으로, 어떤 네트워크들이 활성/억제될 것인지 예측된 결과를 Coronavirus Network Explorer를 통해 확인하는 방법을 알아보았습니다. 현재 COVID-19 관련 연구를 위하여 무료로 사용할 수 있으므로 치료제 개발에 도움이 되길 바라며, 하루빨리 연구가 진척되어 이 사태가 안정화되었으면 합니다.

-
신종 코로나바이러스와 진단키트, https://insilicogen.com/blog/343
-
CLC Genomics Workbench를 이용한 SARS-CoV-2의 MinION 시퀀싱 데이터 분석, https://insilicogen.com/blog/342

-
Gorden et al., 2020. A SARS-CoV-2 Protein Interaction Map Reveals Targets for Drug Repurposing. Nature, Online ahead of print. https://www.nature.com/articles/s41586-020-2286-9
-
Krämer et al., 2013. Causal analysis approaches in Ingenuity Pathway Analysis. Bioinformatics, 30(4):523–530. https://academic.oup.com/bioinformatics/article/30/4/523/202720
작성 : insilico Lab 김형민 컨설턴트
Posted by 人Co
- Response
- No Trackback , No Comment
- RSS :
- https://post-blog.insilicogen.com/blog/rss/response/351
Trackback URL : 이 글에는 트랙백을 보낼 수 없습니다



